No início de 2025, cientistas anunciaram que haviam trazido os lobos-terríveis de volta da extinção com três filhotes de lobo editados geneticamente. O entusiasmo da imprensa pode ter obscurecido alguns detalhes importantes, em especial o fato de que os genomas de lobos-cinzentos existentes foram editados com alterações genéticas específicas de lobos-terríveis. Contudo, o feito demonstrou o potencial de tecnologias genéticas avançadas para reviver genes que se acreditava estarem perdidos para sempre.
Em vez de trazer de volta espécies extintas, no entanto, uma aplicação mais imediata e gerenciável dessa tecnologia é a desextinção molecular, que é a ressurreição seletiva de genes, proteínas ou vias metabólicas extintas. Essa área emergente aproveita duas disciplinas científicas principais: a paleogenômica, o estudo do DNA antigo (aDNA), e a paleoproteômica, a análise de proteínas antigas preservadas em restos fossilizados e subfósseis.
Essas abordagens permitem que os cientistas explorem a história evolutiva em busca de novos compostos bioativos que podem revolucionar a medicina, a biotecnologia e a biologia sintética. Especificamente, a desextinção molecular pode resolver problemas de resistência a antibióticos, fornecendo fontes não convencionais de moléculas antimicrobianas.
Avanços tecnológicos recentes transformaram a desextinção molecular de mera especulação teórica em realidade experimental. O sequenciamento de última geração (NGS) e o sequenciamento de leitura longa de terceira geração melhoraram drasticamente a recuperação de DNA antigo fragmentado, enquanto a espectrometria de massa de alta resolução e a modelagem bioinformática de proteínas permitem que os pesquisadores reconstruam sequências de proteínas antigas e prevejam suas funções.
Com o avanço da biologia computacional e da inteligência artificial, a identificação de moléculas favoráveis evoluiu de um processo amplamente aleatório para uma metodologia mais deliberada e orientada por dados, em que os pesquisadores podem direcionar características moleculares específicas com base em uma análise extensiva de dados para prever sua eficácia potencial.
Tendências de pesquisa em desextinção molecular
Analisamos a CAS Content CollectionTM, o maior repositório de informações científicas com curadoria humana, para compreender o cenário atual e o progresso das pesquisas em desextinção molecular. Descobrimos que os documentos aumentaram nos últimos dez anos, com uma única patente durante esse período (ver figura 1).
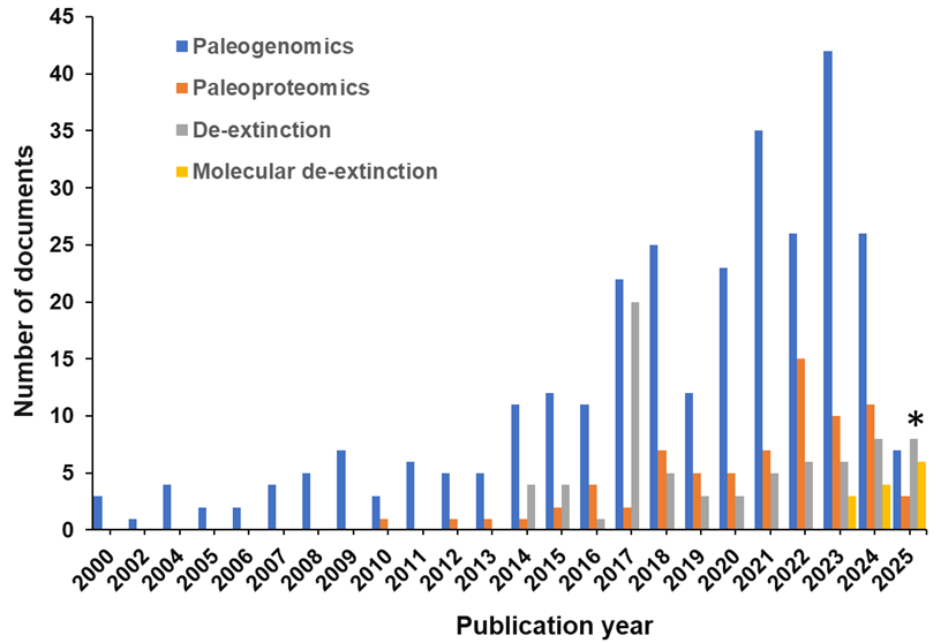
Essa análise revela que o pico de documentos relacionados à desextinção em 2017 está relacionado à publicação de uma edição especial do The Hastings Center for Bioethics, Recreating the Wild: De‐Extinction, Tecnologia, and the Ethics of Conservation. Também examinamos conceitos essenciais relacionados à desextinção na literatura (ver figura 2).
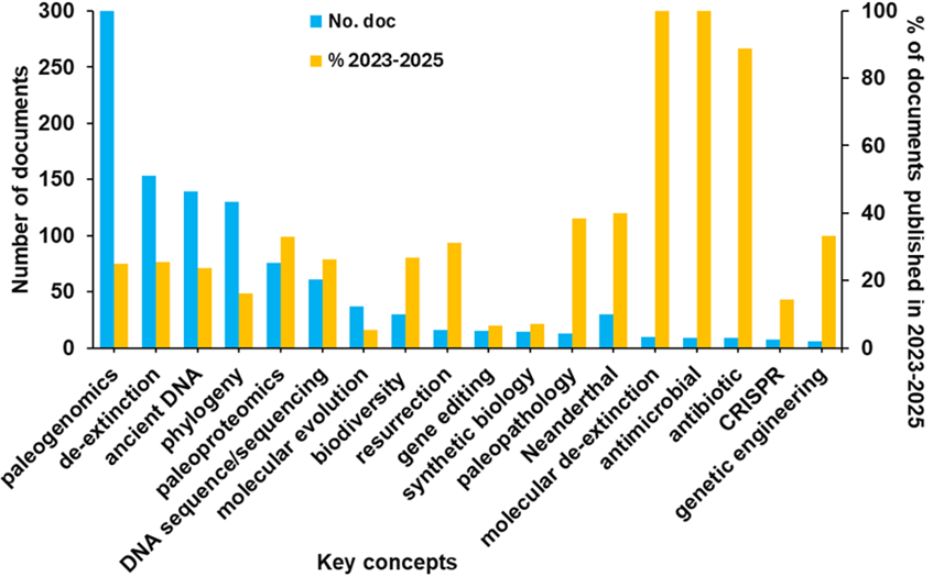
As principais conclusões do cenário de pesquisa atualmente são:
- A paleogenômica é o conceito mais explorado. Enquanto outras abordagens (como back-breeding ou clonagem) contribuem para a desextinção, a paleogenômica fornece o roteiro genético necessário para a ressurreição precisa de espécies. Ao recuperar, sequenciar e analisar o material genético de espécies extintas, os pesquisadores podem reconstruir genomas perdidos, identificar adaptações funcionais essenciais e criar substitutos vivos por meio da edição genômica. Os avanços no sequenciamento de alto rendimento, na edição de genes CRISPR-Cas9 e na biologia sintética estão convergindo para tornar a ressurreição de substâncias e espécies um empreendimento científico tangível, embora complexo.
- Antimicrobianos e antibióticos são o tema da maioria dos documentos relacionados à desextinção molecular nos últimos três anos (considerando dados de 2025 apenas até março). Três interseções principais são dignas de nota: (1) descoberta de peptídeos antimicrobianos antigos por meio da paleogenômica, (2) estratégias antimicrobianas baseadas em CRISPR desenvolvidas a partir de ferramentas de desextinção e (3) descoberta de novos antibióticos a partir de comunidades microbianas ressuscitadas.
- A paleopatologia, o estudo de doenças antigas, ocorre em paralelo com a desextinção. A paleopatologia é tradicionalmente focada na compreensão da saúde em populações antigas por meio de restos esqueléticos e mumificados. No entanto, avanços recentes em paleogenômica, paleoproteômica e bioinformática expandiram suas aplicações para a descoberta de medicamentos modernos. Ao analisar patógenos antigos, respostas imunes humanas e compostos medicinais extintos, os pesquisadores estão descobrindo novas estratégias terapêuticas para combater a resistência aos antibióticos, doenças crônicas e infecções emergentes. Além disso, a integração da biologia evolutiva com a paleopatologia oferece uma melhor compreensão das pressões seletivas que afetaram a suscetibilidade ao câncer em espécies extintas e pode identificar mecanismos potenciais de resistência tumoral.
- Dados em escala genômica sobre neandertais foram disponibilizados a partir de restos esqueléticos de quatorze sítios arqueológicos que abrangem a história dos neandertais em grandes porções de suas áreas geográficas identificadas.
Metodologias de desextinção molecular
Conforme observado, existem duas abordagens em relação à desextinção: a paleogenômica, que estuda DNAs antigos, e a paleoproteômica, que estuda proteínas antigas. Cada um levou a importantes descobertas importantes na identificação de possíveis tratamentos antimicrobianos.
Paleogenômica
A ideia de desextinção deixou de ser ficção científica e se tornou uma busca científica concreta graças aos avanços na paleogenômica. Essa abordagem já produziu insights funcionais sobre a biologia evolutiva, como os mecanismos de adaptação ao frio da megafauna do Pleistoceno, as diferenças neurogenéticas entre os humanos modernos e os neandertais e a evolução do sistema imunológico de patógenos extintos.
A paleogenômica nos ajuda até mesmo a entender nossa resposta aos patógenos atuais. Por exemplo, um estudo dos genes imunológicos dos neandertais explicou nossa suscetibilidade a doenças infecciosas emergentes, como a COVID-19. Um grupo de genes no cromossomo 3, identificado como o principal fator de risco genético para a insuficiência respiratória após a infecção pelo SARS-CoV-2, foi conferido por um segmento genômico herdado dos neandertais e foi encontrado em cerca de 50% das pessoas no sul da Ásia e 16% das pessoas na Europa.
O processo paleogênico visa reviver genes de espécies extintas, reconstruindo seus genomas e introduzindo-os em organismos vivos intimamente relacionados, como no exemplo do lobo-terrível mencionado anteriormente. O primeiro e mais crucial passo é a obtenção de DNA antigo (aDNA) de alta qualidade a partir de material biológico preservado, seguido de isolamento de DNA, sequenciamento de última geração e montagem genética computacional.
Ao contrário do DNA moderno, o aDNA é altamente degradado, quimicamente modificado e frequentemente contaminado com DNA microbiano e ambiental. Avanços na extração de DNA, nas tecnologias de sequenciamento e na bioinformática possibilitaram a recuperação e análise de aDNA, abrindo caminho para os esforços de desextinção.
Trazer de volta animais totalmente formados, como lobos terríveis ou mamutes lanosos, envolve muitos desafios práticos e éticos. O bem-estar animal e as consequências ecológicas são apenas duas das questões complexas relacionadas à desextinção de espécies. Contudo, recuperar peptídeos antigos é muito menos problemático e pode impulsionar os esforços de descoberta de medicamentos. Por meio da paleogenômica, os pesquisadores identificaram oito genomas de vertebrados extintos e os verificaram computacionalmente em busca de defensinas, que são pequenos peptídeos catiônicos ricos em dissulfeto que desempenham funções importantes na imunidade do hospedeiro.
Como resultado, seis β-defensinas autênticas foram identificadas, cinco das quais são derivadas de duas espécies diferentes de aves extintas e uma de mamífero. As análises evolutivas e estruturais dessas moléculas estão abrindo novos caminhos para a descoberta de antibióticos, embora ainda não tenham sido validadas experimentalmente.
Paleoproteômica
A desextinção molecular por meio da paleoproteômica envolve a extração, o sequenciamento, a reconstrução computacional e a ressurreição funcional de proteínas de organismos extintos. Esta metodologia aproveita os avanços em espectrometria de massas, bioinformática e biologia sintética para recuperar e estudar biomoléculas antigas.
Por exemplo, cientistas usaram modelos de aprendizado profundo para descobrir novos peptídeos antibióticos. Os modelos de previsão de sítios proteolíticos foram treinados para projetar a atividade antimicrobiana por uma grande variedade de proteases nos proteomas de organismos extintos (o chamado "extintoma"). Previu-se que uma grande coleção de sequências não encontradas em organismos existentes apresentaria atividade antimicrobiana de amplo espectro. Desses, 69 peptídeos foram sintetizados e sua atividade contra patógenos bacterianos foi verificada experimentalmente. Uma seleção de peptídeos antimicrobianos ativos é apresentada na tabela 1.
| Peptídeo | Organismo extinto | Proteína de origem | Sequência de peptídeo | MIC |
|---|---|---|---|---|
| Hydrodamin-1 3078251-51-8 |
Dugongo-de-steller (Hydrodamalis gigas) | Gene 1 de diferenciação endotelial | LYCRIYSLVRARG RRLTFRKNISK | 4 μmol L-1 (A. baumannii, E. faecium) |
| Megalocerin-1 3078251-56-3 |
Alce-gigante (Megaloceros gigantescus) | Subunidade 3 da citocromo c oxidase | LIVCFFRQLKFHF | 8 μmol L-1 (A. baumannii, E. faecium) |
| Mylodonin-2 3078251-68-7 |
Preguiça-gigante (Mylodon darwinii) | Apolipoproteína B | KRKRGLKLATALS LNNKF | 32 μmol L-1 (E. coli) |
| Elephasin-2 3078251-59-6 |
Elefante-de-presas-retas (Elephas antiquus) | Subunidade 8 da ATP sintase F0 | IFLHLKILKIIRLL | 1 μmol L-1 (A. baumannii, S. aureus, E. faecium) |
| Mammuthusin-2 3078251-49-4 |
Mamute-lanoso siberiano (Mammuthus primigenius) | Receptor de melanocortina-1 | RACLHARSIARLHK RWRPVHQGLGLK | 32 μmol L-1 (A. baumannii, E. faecium) |
| Equusin-1 3078251-35-8 |
Zebra-de-grant (Equus quagga boehmi) | Proteína 1 associada à resistência natural de macrófagos) | FLKLRWSRFARVLL | 1 μmol L-1 (E. faecium) 4 μmol L-1 (A. baumannii, E. coli, P. aeruginosa) |
| Equusin-2 3078251-48-3 |
Zebra-de-grant (Equus quagga boehmi) | Proteína associada à microcefalia do tipo fusiforme anormal | KIYKKLSTPPFTL NIRTLPKVKFPK | 8 μmol L-1 (A. baumannii) |
Diversos pares de peptídeos do mesmo organismo extinto exibiram fortes interações sinérgicas contra patógenos como A. baumannii e P. aeruginosa, com valores de índice de concentração inibitória fracionária (FIC) tão baixos quanto 0,38 para A. baumannii. Para a combinação de Equusin-1 e Equusin-3, as concentrações inibitórias mínimas (MICs) diminuíram 64 vezes (de 4 μmol L-1 para 62,5 nmol L-1), atingindo concentrações submicromolares comparáveis às MICs dos antibióticos mais potentes.
Notavelmente, os principais compostos, incluindo Mammuthusin-2, Elephasin-2, Hydrodamin-1, Mylodonin-2 e Megalocerin-1, exibiram potencial atividade anti-infecciosa em camundongos com abscesso cutâneo ou infecções na coxa. Os resultados obtidos para os peptídeos mais ativos testados no modelo de infecção por abscesso cutâneo (Elephasin-2 e Mylodonin-2) indicaram uma atividade antibacteriana comparável à do antibiótico polimixina B, amplamente utilizado. Da mesma forma, o Mylodonin-2 e o Elephasin-2 apresentaram eficácia anti-infecciosa comparável à da polimixina B ao usar um modelo de infecção profunda da coxa de camundongos, ressaltando o potencial da desextinção molecular como uma abordagem bem-sucedida na descoberta de antibióticos.
Uma patente recente revelou os métodos para identificar peptídeos antimicrobianos derivados de proteomas extintos usando um algoritmo de aprendizado profundo multitarefa, APEX, juntamente com os 41 peptídeos antimicrobianos identificados e sua sinergia e mecanismo de ação.
Além disso, a ferramenta de aprendizado de máquina criada, o modelo de floresta aleatória panCleave para a previsão de locais de clivagem em todo o proteoma, explorou um classificador de locais de clivagem de pan-proteases para realizar a proteólise computacional — uma digestão in silico de proteínas humanas. As abordagens de aprendizado de máquina foram, portanto, usadas para a desextinção molecular e exploraram os proteomas dos nossos parentes mais próximos, os humanos arcaicos neandertais e denisovanos. Vários antibióticos peptídicos criptografados foram ressuscitados, os quais exibiram atividade antimicrobiana in vitro e em modelos pré-clínicos de camundongos.
Possíveis descobertas importantes em antibióticos
Para entender como pode ter sido o ancestral dos antibióticos glicopeptídicos atualmente, os pesquisadores usaram bioinformática e métodos genéticos e bioquímicos para trazer a "paleomicina" ancestral de volta à vida. Primeiro, foi prevista a linha de montagem da sintetase peptídica não ribossômica da paleomicina e foi construída uma árvore guia com base em agrupamentos de genes biossintéticos. Posteriormente, empregando técnicas de biologia sintética, os pesquisadores reconstruíram o peptídeo previsto e validaram sua atividade antibiótica.
Esse estudo comprovou que a combinação de biologia sintética e técnicas de computação pode determinar a evolução temporal dos antibióticos e, possivelmente, ressuscitar moléculas antigas. Também demonstrou as táticas de otimização natural alcançadas por meio de processos evolutivos que resultaram em antibióticos glicopeptídicos modernos, estabelecendo, assim, a base para esforços futuros de engenharia dessa importante classe de agentes antimicrobianos.
Os cientistas também estão explorando a possibilidade de encontrar novas fontes de antibióticos nas catelicidinas de neandertais. Trata-se de uma família de peptídeos antimicrobianos semelhantes aos encontrados em humanos modernos, que desempenham um papel na defesa contra infecções. Os pesquisadores desenvolveram um modelo de aprendizado de máquina que pode extrair dados proteômicos e genômicos de neandertais e denisovanos, encontrando, essencialmente, sequências de humanos arcaicos e prevendo quais seriam candidatos viáveis a antibióticos.
Desafios e oportunidades na desextinção
A desextinção molecular pode apresentar menos dilemas logísticos e éticos do que ressuscitar animais extintos e reintroduzi-los nos ecossistemas modernos. No entanto, ela não está livre de certos riscos, como:
- A degradação do DNA e os dados genômicos incompletos dificultam a reconstrução completa dos genes.
- Incerteza funcional de moléculas ressuscitadas, incluindo erros de dobramento de proteínas, modificações pós-traducionais, toxicidade e imunogenicidade.
- Silenciamento de genes, efeitos colaterais e transferência horizontal de genes, em que os genes modificados podem se espalhar de forma incontrolável nos ecossistemas e causar impactos não intencionais.
- Questões bioéticas sobre se as moléculas extintas devem ser comercializadas.
As estruturas éticas e a colaboração entre as comunidades científicas e regulatórias serão vitais para orientar muitas dessas considerações. Em termos de possíveis obstáculos científicos, as tecnologias avançadas continuarão desempenhando um papel fundamental. A IA pode simular como as proteínas se dobram e funcionam, dispensando a necessidade de sequências completas de DNA. As redes neurais podem prever fragmentos ausentes em DNAs antigos degradados e melhorar a precisão da reconstrução. O CRISPR-Cas9 e a edição de bases podem, potencialmente, "humanizar" genes antigos para uso médico seguro.
A desextinção molecular representa uma mudança de paradigma na descoberta de antibióticos, oferecendo um reservatório singular de potencial antimicrobiano inexplorado. Embora ainda haja desafios em termos de escala e regulamentação, os primeiros sucessos demonstram que a biodiversidade perdida da Terra pode ser a chave para solucionar a crise da resistência antimicrobiana.
Para mais informações, consulte nosso artigo na ACS Omega, Molecular Paleontology Meets Drug Discovery: The Case for De-extinct Antimicrobials.
Referências:
2: https://www.nature.com/articles/s41551-024-01201-x?utm_source=acs&getft_integrator=acs