A transcriptômica espacial representa um avanço considerável na pesquisa biológica, oferecendo uma técnica poderosa para examinar a complexa relação entre a expressão gênica e a organização espacial dentro de um tecido intacto. Revelando o arranjo espacial dos componentes celulares e seus estados transcricionais, esse método permite uma compreensão mais profunda da composição e atividade celular em sistemas biológicos complexos.
A transcriptômica espacial abrange vários métodos que compartilham o objetivo de atribuir dados transcriptômicos à localização original das moléculas de RNA dentro do tecido. Este avanço tecnológico oferece insights sem precedentes sobre a organização, a função e as interações das células em tecidos complexos. Devido ao seu potencial de melhorar vários campos biológicos e médicos, como biologia do desenvolvimento, neurociência e pesquisa do câncer, a transcriptômica espacial foi reconhecida como “Método do Ano” pela Nature Methods em 2020.
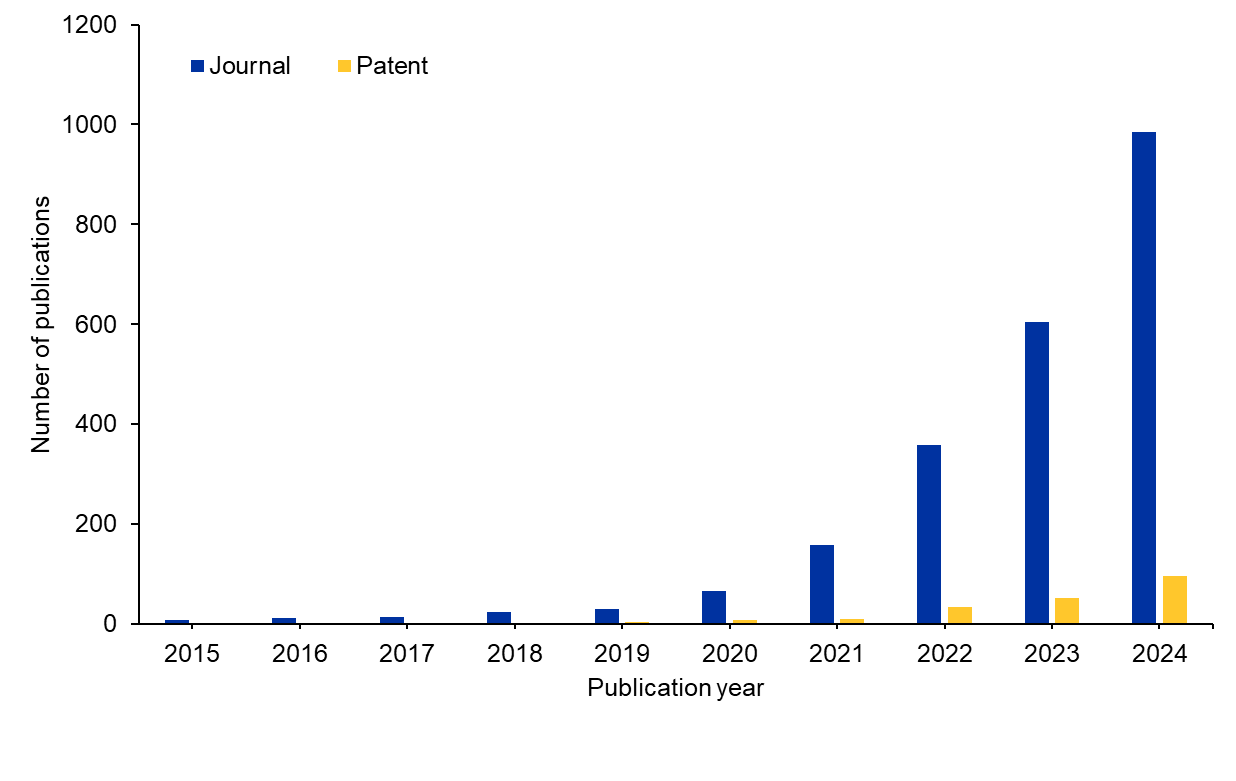
Há outras abordagens transcriptômicas na biomedicina, mas recentemente os pesquisadores fizeram avanços animadores com transcriptômica espacial. Examinamos a Coleção de Conteúdo do CASTM, o maior repositório de informações científicas com curadoria por humanos, para conhecer melhor o panorama da pesquisa da transcriptômica espacial na última década. Observamos um enorme aumento (cerca de 150 vezes) nas publicações relacionadas à transcriptômica espacial nos últimos dez anos (ver Figura 1). As publicações em periódicos dominam a literatura em comparação com as publicações de patentes, sugerindo que ainda há grande oportunidade de comercialização.
Como a ciência da transcriptômica está mudando e o que isso significa para novas abordagens de tratamento para várias doenças? Vamos explorar como funciona a transcriptômica espacial e os rumos da pesquisa.
O que é transcriptômica espacial?
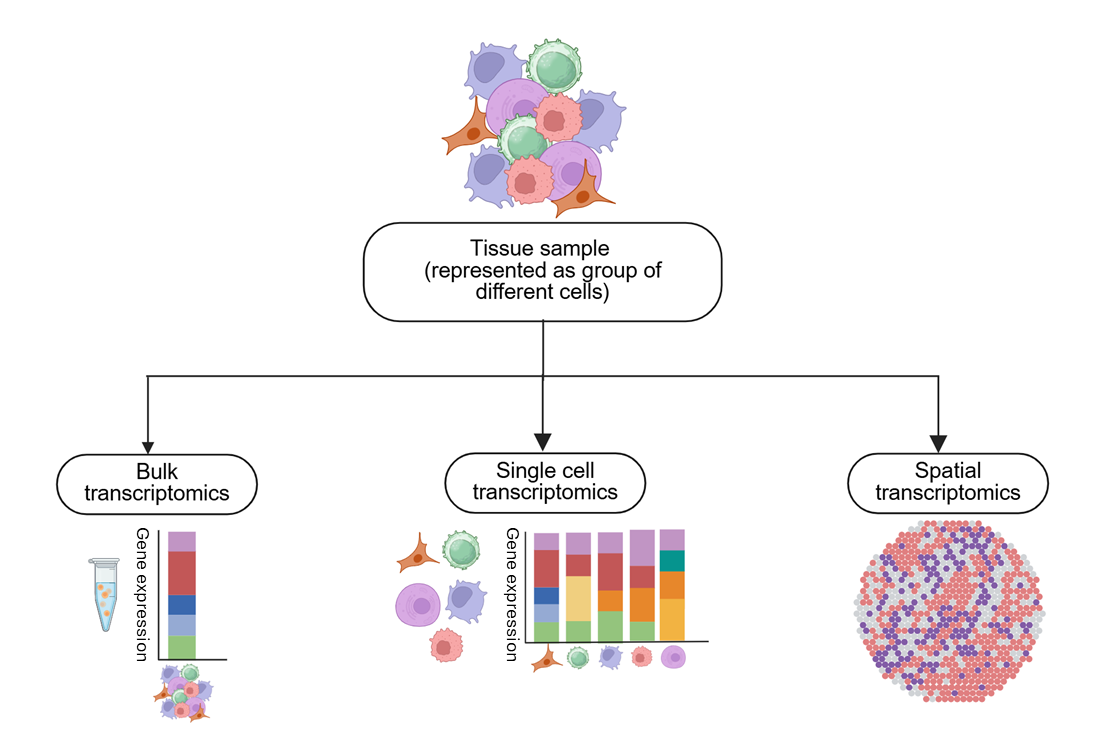
Há atualmente três tipos principais de transcriptômica: em massa, de célula única e espacial (veja a Figura 2). A transcriptômica em massa analisa a expressão gênica em uma mistura de células, fornecendo uma visão global, mas sem resolução no nível de uma única célula. A transcriptômica de célula única resolve a expressão gênica em células individuais, permitindo insights sobre a heterogeneidade celular e populações de células raras, mas perde o contexto espacial.
A transcriptômica espacial preserva o local da expressão gênica nos tecidos, revelando interações celulares e a arquitetura do tecido, embora deva ser observado que essa técnica requer uma análise computacional sofisticada. Juntas, essas abordagens oferecem visões complementares da regulação gênica, cada uma delas adequada a diferentes questões de pesquisa.
Uma das primeiras técnicas de transcriptômica espacial foi a FISH (hibridização fluorescente in situ), que visualiza diretamente as moléculas de RNA dentro de células e tecidos intactos. Ele funciona projetando sondas curtas de DNA ou RNA marcadas com fluorescência, complementares à sequência de RNA alvo. Essas sondas hibridizam com o RNA alvo na amostra, e as localizações dos sinais fluorescentes são detectadas com microscopia de fluorescência.
Técnicas como FISH de molécula única (smFISH) melhoraram a sensibilidade, permitindo a detecção e a quantificação de transcritos de RNA individuais como pontos limitados por difração. As aplicações iniciais da FISH só podiam visualizar a distribuição espacial de um pequeno número de mRNAs bem caracterizados, devido à sobreposição espectral dos fluoróforos. Essas técnicas iniciais também eram demoradas e trabalhosas, especialmente na análise de grandes seções de tecido ou muitos alvos. Devido a essas limitações, os pesquisadores desenvolveram métodos modernos com maior eficiência.
Os avanços tecnológicos melhoram a resolução e a análise
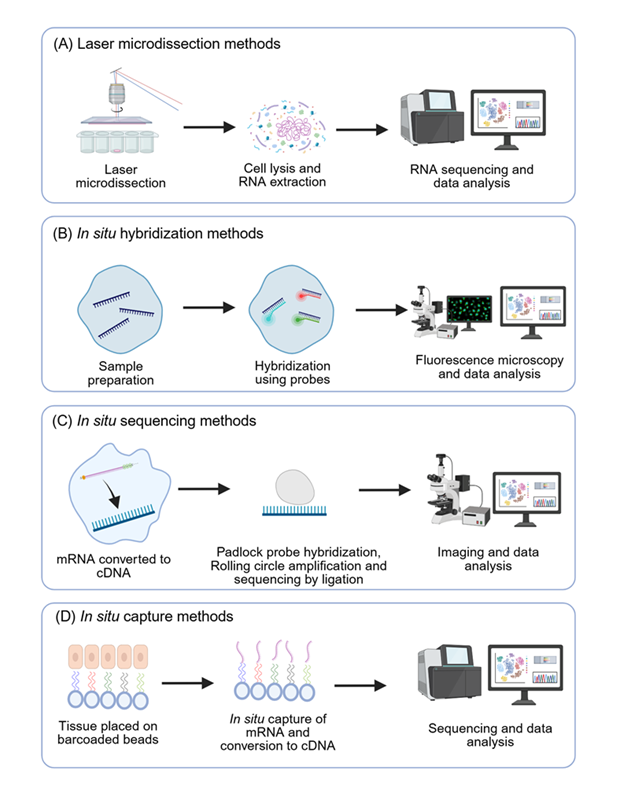
As tecnologias de transcriptômica espacial podem ser divididas em quatro categorias: métodos de microdissecção, métodos de hibridização in situ (ISH), métodos de sequenciamento in situ (ISS) e métodos de captura in situ (ISC) (consulte a Figura 3). A escolha da tecnologia geralmente depende de objetivos específicos de pesquisa, como a necessidade de alta resolução, ampla cobertura de genes ou análise direcionada. Como vimos nos números de publicações da Coleção de Conteúdo do CAS, esse campo passou por uma rápida evolução tecnológica na última década. Esses avanços possibilitam que os pesquisadores identifiquem insights críticos sobre os mecanismos da doença e desenvolvam possíveis estratégias de tratamento.
Métodos de microdissecção por captura a laser
A Microdissecção por captura a laser (LMD) fornece um meio físico para isolar populações celulares específicas ou regiões de interesse diretamente de seções de tecido sob visualização microscópica. Depois que as áreas desejadas são microdissecadas, o RNA é extraído dessas células isoladas ou fragmentos de tecido e, em seguida, analisado por meio do sequenciamento tradicional de RNA em massa (RNA-seq). Essa abordagem vincula os perfis de expressão gênica a locais espaciais específicos definidos pela microdissecção.
Embora o LMD se concentre no isolamento espacial e não na análise in situ, sua integração com tecnologias avançadas de RNA-seq aumentou consideravelmente sua utilidade na transcriptômica espacial. Melhorias na extração de RNA de um pequeno número de células, métodos de amplificação e profundidade de sequenciamento permitiram uma análise mais abrangente do transcriptoma de amostras microdissecadas. Os instrumentos de LMD mais recentes oferecem maior precisão e automação, tornando o processo mais eficiente. Eles podem ser cruciais para compreender a heterogeneidade molecular dentro das amostras de tecido, identificando populações de células específicas que estão causando doenças e, possivelmente, orientando terapias direcionadas.
Métodos ISH
ISH abrange muitas técnicas que usam sondas marcadas para detectar sequências de ácido nucleico específicas (tanto DNA quanto RNA) em células ou tecidos, preservando seu contexto espacial. Semelhante ao FISH, o ISH depende da hibridização de sondas marcadas às sequências-alvo. No entanto, o ISH pode empregar vários métodos de detecção além da fluorescência, incluindo marcadores enzimáticos ou radioativos, que podem ser visualizados por meio de microscopia ou autorradiografia.
A ISH tradicional com detecção cromogênica (por exemplo, com enzimas que produzem um precipitado colorido) tem sido uma técnica fundamental na biologia do desenvolvimento e na histologia há décadas, permitindo a visualização dos padrões de expressão gênica na resolução celular. A ISH radioativa oferece maior sensibilidade para transcritos de baixa abundância. A FISH é comparável a um subconjunto de ISH baseado em fluorescência.
A evolução do ISH tem se concentrado principalmente no aumento da sensibilidade, da capacidade de multiplexação e da resolução. FISH multiplexada recente, como MERFISH, seqFISH e outras variantes, permite a detecção simultânea de centenas a milhares de mRNAs na mesma amostra, empregando marcação combinatória e rodadas sequenciais de hibridização e imagem.
Métodos ISS
Esses métodos geralmente envolvem a conversão de RNA em DNA complementar (cDNA) dentro da seção de tecido, seguida por amplificação in situ e sequenciamento com várias estratégias de codificação. As informações de sequência são então decodificadas por meio de rodadas iterativas de hibridização, ligação ou reações enzimáticas, combinadas com imagens de alta resolução. Os dados resultantes fornecem a identidade e a localização espacial de vários transcritos de RNA em resolução subcelular.
São exemplos de tecnologias ISS sequenciamento miniaturizado de RNA total de célula única (STORM-seq), sequenciamento fluorescente in situ (FISSEQ) e métodos de sequenciamento baseados em sondas padlock. Essas técnicas podem alcançar alta multiplexação, permitindo a detecção simultânea de milhares de espécies de RNA em células individuais em seu contexto espacial nativo.
Métodos ISC
Os métodos de ISC envolvem o posicionamento de oligonucleotídeos com identificadores espaciais em uma seção de tecido para capturar o mRNA liberado pelas células. As informações espaciais são codificadas nos identificadores exclusivos nas sondas de captura. Após a captura do mRNA e a transcrição reversa, o cDNA resultante é agrupado e sequenciado usando o sequenciamento padrão de próxima geração. Ao decodificar os identificadores espaciais associados a cada molécula de cDNA, o perfil de expressão gênica pode ser mapeado de volta à sua localização original no tecido.
|
Resolution (resolução) |
Sensibilidade |
Capacidade de processamento |
Custo |
|
|
Microdissecção |
Alto |
Alto |
Baixo |
Moderado |
|
Hibridização in situ |
Baixo |
Moderado |
Alto |
Baixo a |
|
Sequenciamento in situ |
Alto |
Alto |
Alto |
Alto |
|
Captura in situ |
Alto |
Alto |
Alto |
Moderado a |
Tabela 1: Comparação entre diversas tecnologias de ST
A tecnologia original de transcriptômica espacial, desenvolvida no Karolinska Institute, é um excelente exemplo de um método ISC, comercializado posteriormente como a plataforma 10x Genomics Visium. Utiliza microarrays com identificador espacial para capturar mRNA. As inovações subsequentes incluem o Slide-seq, que usa esferas com identificadores de DNA depositados em uma lâmina.
Os métodos iniciais de ISC tinham resolução espacial relativamente baixa, limitada pelo tamanho dos pontos ou esferas de captura. Foram feitos progressos consideráveis no aumento da densidade de identificadores espaciais, na redução da área de captura e na melhoria da eficiência da captura de mRNA e da síntese de cDNA. Técnicas como transcriptômica espacial de alta definição (HDST) e Slide-seqV2 aumentaram drasticamente a resolução, aproximando dos níveis de célula única em alguns casos.
Os métodos ISC oferecem uma abordagem escalável e relativamente econômica para o perfil espacial do transcriptoma completo, tornando-os atraentes para estudos em larga escala em pesquisa básica, desenvolvimento de medicamentos e possivelmente diagnósticos clínicos. A capacidade de analisar todo o transcriptoma com contexto espacial oferece uma visão abrangente da organização e função do tecido.
A transcriptômica espacial melhora nossa compreensão da biologia das doenças e da descoberta de medicamentos
A compreensão do contexto espacial da expressão gênica fornece insights essenciais para desvendar os mecanismos das doenças. Isso também afeta a descoberta e o desenvolvimento de medicamentos. Atualmente, as quatro áreas de foco da transcriptômica espacial são o câncer, a neurociência, a biologia do desenvolvimento e as doenças infecciosas.
Câncer
Um dos desafios mais significativos na pesquisa sobre o câncer tem sido compreender e abordar a heterogeneidade do tumor e a complexa interação entre os tumores e o microambiente circundante (TME). O TME é uma entidade complexa e em constante evolução que normalmente envolve células imunológicas, células estromais, vasos sanguíneos e uma matriz extracelular. Graças aos avanços na transcriptômica espacial, a função vital do microambiente imunológico é agora mais conhecida, revelando os vários mecanismos que as células cancerosas usam para contornar o sistema imunológico do hospedeiro.
Agora é amplamente aceito que o TME é um promotor ativo da progressão do câncer e não apenas uma entidade neutra. Ele regula direta ou indiretamente o crescimento do câncer, a metástase, a resistência ou a suscetibilidade a vários tratamentos. Como as técnicas de transcriptômica espacial preservam o contexto espacial, são ferramentas excelentes para estudar as interações entre tumor-TME e células tumorais-imunes e fornecem novos insights sobre nosso conhecimento da biologia tumoral.
Houve vários estudos notáveis recentemente. Por exemplo, no glioblastoma foi demonstrado que as células mieloides liberadoras de IL-10 (localizadas em regiões tumorais semelhantes ao mesênquima) levam à exaustão das células T, contribuindo para o TME imunossupressor. No carcinoma espinocelular oral, arquiteturas distintas e conservadas do núcleo e da borda do tumor foram identificadas e caracterizadas, o que previu a sobrevivência e a resposta terapêutica direcionada. A transcriptômica espacial também é amplamente utilizada na pesquisa do câncer de mama para estudar a heterogeneidade tumoral e a resposta a medicamentos impulsionada pelo TME.
[Adicionar separador]: Os inibidores de PPI são promissores no combate ao câncer [link]
Neurociência
A notável complexidade funcional do cérebro decorre da disposição espacial precisa e das conexões entre as diversas populações de células neuronais e gliais. A transcriptômica espacial tem a capacidade de dissecar essa organização no nível celular e apresentar insights sobre como os circuitos neuronais se formam e se tornam funcionais, como as diversas regiões do cérebro são especificadas e como as interrupções dessa arquitetura bem ajustada podem levar ao início e/ou progressão de vários distúrbios neurológicos.
A transcriptômica espacial agora é usada para decifrar mecanismos críticos e agentes moleculares na patologia da placa amiloide, uma característica marcante do Alzheimer. Em modelos de camundongos, os cientistas identificaram redes de coexpressão gênica envolvidas na fase inicial versus na fase tardia da doença (genes de mielina e oligodendrócitos [OLIGs] e genes induzidos por placa [PIGs], respectivamente). Em outro estudo, foram identificadas associações entre os perfis regionais de expressão gênica e a vulnerabilidade regional seletiva às patologias amiloide-beta e tau em pacientes com Alzheimer.
A transcriptômica espacial também nos ajuda a descobrir possíveis alvos moleculares que poderiam melhorar as imunoterapias direcionadas ao beta-amiloide. Os pesquisadores estão estudando os mecanismos neuroimunes envolvidos na eliminação de placas de beta-amiloide em pacientes que receberam imunização ativa ou passiva (regulação positiva de genes associados ao eixo TREM2-APOE). Além disso, foi utilizada uma nova estrutura computacional para integrar "dados de transcriptômica espacial com autoencoders baseados em grafos superparametrizados com dados de imagem de cromatina (STACI)" para identificar possíveis novos biomarcadores de Alzheimer.
Além da pesquisa sobre Alzheimer, a transcriptômica espacial apresentou novos insights sobre vários genes de risco e disregulações de vias na esclerose lateral amiotrófica (ELA).
Biologia do desenvolvimento
Vários estudos destacaram o poder da transcriptômica espacial na biologia do desenvolvimento. Aplicando o método Stereo-seq para gerar o atlas transcriptômico espaço-temporal da organogênese de camundongos (MOSTA), os pesquisadores obtiveram insights valiosos sobre a base molecular da heterogeneidade espacial das células e da especificação do destino celular em tecidos em desenvolvimento, como o mesencéfalo dorsal. Em outro estudo, a transcriptômica espacial foi usada para perfilar padrões de expressão gênica espaço-temporal na cardiogênese humana e mapear a distribuição de tipos celulares e a organização espacial no coração embrionário humano.
Em outro desenvolvimento, um atlas transcriptômico completo do cérebro de rato foi gerado com informações espaciais para 308 clusters de células em resolução de célula única, envolvendo mais de 4 milhões de células. Isso proporciona um recurso valioso para estudar o desenvolvimento, a função e a regulação gênica do cérebro de camundongos.
Doenças infecciosas
A transcriptômica espacial está transformando o estudo de doenças infecciosas ao proporcionar a capacidade de analisar as intrincadas interações entre o hospedeiro e o patógeno no contexto espacial dos tecidos infectados. Esta metodologia tem sido empregada com sucesso para estudar infecções bacterianas como Mycobacterium tuberculosis, Pseudomonas aeruginosa, e Echinococcus multilocularis. Também é usado em pesquisas sobre infecções virais, como SARS-COV-2 e HIV. Os pesquisadores identificaram diretamente certos padrões de co-localização de transcritos do hospedeiro e do patógeno e descobriram fatores do hospedeiro que modulam a resposta imune.
Desafios e oportunidades na transcriptômica espacial
Embora as abordagens transcriptômicas espaciais ofereçam insights excepcionais, também apresentam alguns desafios. Os dados gerados pela maioria dos métodos de transcriptômica espacial são enormes, complexos e às vezes ruidosos devido à baixa eficiência de captura de RNA, vieses de amplificação por PCR ou erros de sequenciamento. Isso geralmente exige algoritmos sofisticados para processamento de imagens, mapeamento espacial e integração com sequenciamento de RNA de célula única. A fixação, seção e a coloração dos tecidos podem introduzir defeitos que afetam a integridade do RNA e a resolução espacial. As técnicas de manuseio e processamento precisam ser otimizadas para minimizar esses problemas.
Os reagentes, o sequenciamento e a infraestrutura computacional necessários para a transcriptômica espacial podem ser caros, limitando a disponibilidade dessas inovações. Além disso, essas abordagens envolvem fluxos de trabalho experimentais complexos, exigindo pesquisadores com experiência em biologia molecular, bioinformática e microscopia. Habilidades interdisciplinares são essenciais para uma análise e interpretação precisas. Além disso, os protocolos de transcriptômica espacial variam consideravelmente entre os laboratórios, o que dificulta a comparação dos resultados entre os estudos.
Apesar desses desafios, o campo da transcriptômica espacial continua a evoluindo rapidamente, com esforços contínuos dedicados à melhoria da resolução espacial, no aumento da sensibilidade e no aumento do rendimento. Outra possibilidade é a integração com outros dados ômicos, conhecida como multiômica espacial, que poderia proporcionar conhecimentos mais abrangente dos processos biológicos e patológicos combinando genômica, proteômica, epigenômica e metabolômica — tudo dentro de uma estrutura espacial.
Os avanços constantes estão melhorando os métodos de análise de dados e reduzindo o ruído para uma melhor compreensão dos dados de ST. Como muitos outros campos biomédicos, a IA está pronta para revolucionar a transcriptômica espacial ao tornar a análise de dados mais eficiente e perspicaz. Alguns exemplos recentes são Path2space, modelo de aprendizado profundo projetado para prever a expressão gênica espacial diretamente de lâminas de histopatologia de biópsias de câncer de mama e prever a resposta dos pacientes à quimioterapia e ao trastuzumabe, e Spotiphy, abordagem baseada em aprendizado de máquina para alcançar resolução de célula única e cobertura de todo o genoma. As metodologias de transcriptômica espacial in vivo ainda estão em seus estágios iniciais, mas em breve serão alcançáveis graças aos esforços constantes de pesquisas.
O futuro da transcriptômica espacial é promissor. Está preparada para impulsionar novas descobertas inovadoras na pesquisa básica e ter impacto cada vez maior na medicina translacional.