
As fronteiras da química sintética estão sendo levadas a seus limites, fazendo com que os pesquisadores busquem soluções na biologia. Com o surgimento da biologia sintética e da inteligência artificial (IA), os cientistas estão liberando novas possibilidades para a descoberta de medicamentos, como projetar e produzir moléculas raras e inéditas na natureza. Ao aproveitar essas ferramentas, os pesquisadores estão desenvolvendo novas abordagens para sintetizar moléculas complexas, otimizar os caminhos de produção e projetar sistemas biológicos para inovação farmacêutica. Para obter insights sobre esse campo em rápida evolução, conversamos com Graham Hudson, Doutor, especialista em biologia sintética e microbiologia, e Nathan Lanclos, especialista em projeto de proteínas orientado por IA. Saiba mais sobre como o CAS está enfrentando esses desafios.
CAS: A biologia sintética é uma etapa necessária para a inovação no desenvolvimento de medicamentos além da química sintética?
Teoricamente, desde que uma estrutura seja quimicamente perfeita, a química sintética pode ter acesso a ela. Mas a questão não é apenas sobre viabilidade — é sobre eficiência, custo e escalabilidade. Há compostos que, em teoria, você poderia sintetizar, mas o esforço necessário para desenvolver uma rota e a grande dificuldade de controlar a seletividade tornam isso impraticável. É aí que a biologia sintética pode realmente fornecer um novo caminho para a síntese de medicamentos.
A natureza passou milhões de anos refinando o maquinário enzimático capaz de construir essas moléculas complexas com precisão notável, muitas vezes alcançando regio- e estére- seletividade, que podem ser incrivelmente difíceis de obter com a química sintética. Se você pegar algo como o paclitaxel, uma molécula densa em estereocentros e grupos funcionalmente semelhantes, torna-se incrivelmente difícil manipulá-la quimicamente com a precisão necessária para garantir a formação do produto correto. Mas, ao cooptar as ferramentas biossintéticas da natureza, podemos aproveitar as enzimas que montam essas moléculas de forma eficiente.
Além disso, podemos levar isso adiante com o surgimento da biologia computacional e do aprendizado de máquina. As ferramentas de IA nos permitem projetar enzimas além do que a evolução produziu, modificando suas estruturas para catalisar novas reações ou expandindo seu escopo de substratos. Isso significa que não estamos apenas recriando produtos naturais — estamos abrindo a porta para novas moléculas na natureza que podem ter propriedades biológicas totalmente novas.
CAS: Quais descobertas importantes estão ajudando cientistas a transformar micróbios em "fábricas vivas de medicamentos"?
Microrganismos como Saccharomyces (levedura) e Streptomyces (bactérias) já são amplamente utilizados para produzir compostos valiosos, desde antibióticos até agentes anticancerígenos. O principal desafio tem sido otimizar esses sistemas para produzir altos rendimentos de forma eficiente. Os avanços em IA, triagem de alto rendimento e modelagem computacional nos permitem projetar e otimizar vias biossintéticas em uma escala sem precedentes.
Agora podemos usar o aprendizado de máquina para prever como as vias metabólicas se comportarão, permitindo-nos ajustar os níveis de expressão das enzimas, otimizar a disponibilidade de precursores e até modificar o metabolismo do hospedeiro para maximizar a produção. Por exemplo, há muito tempo é difícil projetar experimentos em grandes complexos de proteínas heteroméricas para entender profundamente as interações proteína-proteína de longo alcance, mas agora estamos encontrando maneiras de fazer isso com uma granularidade cada vez maior.
Um exemplo perfeito de como a biologia sintética está transformando a produção de medicamentos é o QS-21 (Stimulon, abaixo), um adjuvante imunoestimulante usado em vacinas. Historicamente, o QS-21 tem sido extraído da árvore de casca de sabão (Quillaja saponaria), mas o processo apresenta vários desafios importantes. Os rendimentos dessas árvores são incrivelmente baixos, tornando a produção em larga escala ambientalmente insustentável e economicamente impraticável. Além disso, depender da extração de plantas significa que as cadeias de suprimentos são vulneráveis à variabilidade climática, desmatamento e fatores geopolíticos.
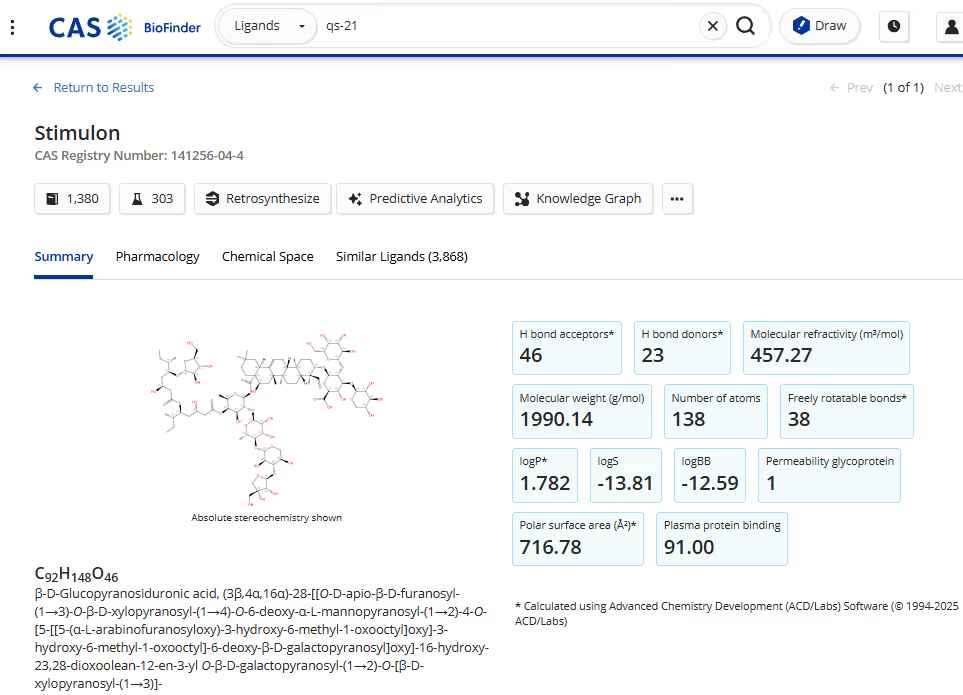
Antes mesmo de tentarmos projetar a levedura para produzir QS-21, primeiro tivemos que mapear toda a sua via biossintética e identificar os genes responsáveis por sua síntese. Este processo foi um esforço hercúleo liderado pelo laboratório de Anne Osbourn no John Innes Centre, que envolveu uma análise transcriptômica minuciosa em diferentes estágios do ciclo de crescimento da planta.
Ao contrário dos agrupamentos de genes biossintéticos microbianos (que tendem a estar localizados no genoma), as vias das plantas estão dispersas em vários cromossomos, frequentemente com sequências não codificantes entre os genes. Essa dispersão tornou a descoberta da via muito mais complexa, exigindo estudos meticulosos de correlação transcriptômica para identificar quais genes estavam funcionalmente envolvidos na biossíntese do QS-21.
CAS: Quais foram os aspectos mais difíceis da adaptação das vias biossintéticas derivadas de plantas para a produção microbiana?
Um dos maiores desafios foi a biossíntese do açúcar. O QS-21 faz parte da classe de produtos naturais denominados saponinas, que são triterpenoides glicosilados complexos. Tem sete açúcares diferentes ligados à sua estrutura central, mas a levedura produz naturalmente apenas um deles. Isso significa que, além de inserir o caminho biossintético para o QS-21, também tivemos que projetar a levedura para sintetizar açúcares não nativos.
Outro problema importante era a toxicidade. Saponinas como o QS-21 são compostos ativos na membrana que podem romper as bicamadas lipídicas e sequestrar esteróis nativos essenciais, o que é, em parte, o que os torna adjuvantes eficazes. No entanto, isso criou um problema considerável na levedura — as moléculas de QS-21 comprometeram a integridade da membrana celular da levedura, levando à morte da célula e à falha na produção. Para resolver isso, tivemos que modificar a composição do esterol da levedura para evitar a ruptura da membrana e otimizar as condições de produção.
Todo esse esforço demonstra o poder da biologia sintética. Sem esses avanços, a produção de QS-21 continuaria limitada pela extração de plantas. Com cepas de bioprodução projetadas, agora temos uma maneira escalável, controlada e sustentável de produzir QS-21. Embora seja necessária mais otimização antes que a produção em grande escala seja viável, esse trabalho abre caminho para uma formulação de vacinas mais eficiente.
CAS: Em quais outras áreas você está aproveitando a IA para acelerar descobertas?
A IA nos permite projetar e otimizar as vias biossintéticas de uma forma que seria impossível apenas por meio de métodos tradicionais. Em vez de tentativa e erro, podemos usar modelos preditivos para determinar quais modificações nas enzimas melhorarão a atividade, quais cepas hospedeiras serão mais compatíveis e como os diferentes componentes da via irão interagir.
Por exemplo, no Keasling Lab, usamos IA para projetar sintases de policetídeos (PKSs), que produzem uma ampla variedade de compostos bioativos, de antibióticos a medicamentos anticâncer. As enzimas PKS são enormes, com módulos individuais contendo milhares de aminoácidos. Seu tamanho e complexidade tornam as abordagens tradicionais de engenharia de proteínas incrivelmente difíceis. A IA nos permite modelar essas proteínas maciças, prever como as modificações afetam a função e projetar novas variantes de enzimas com atividade aprimorada.
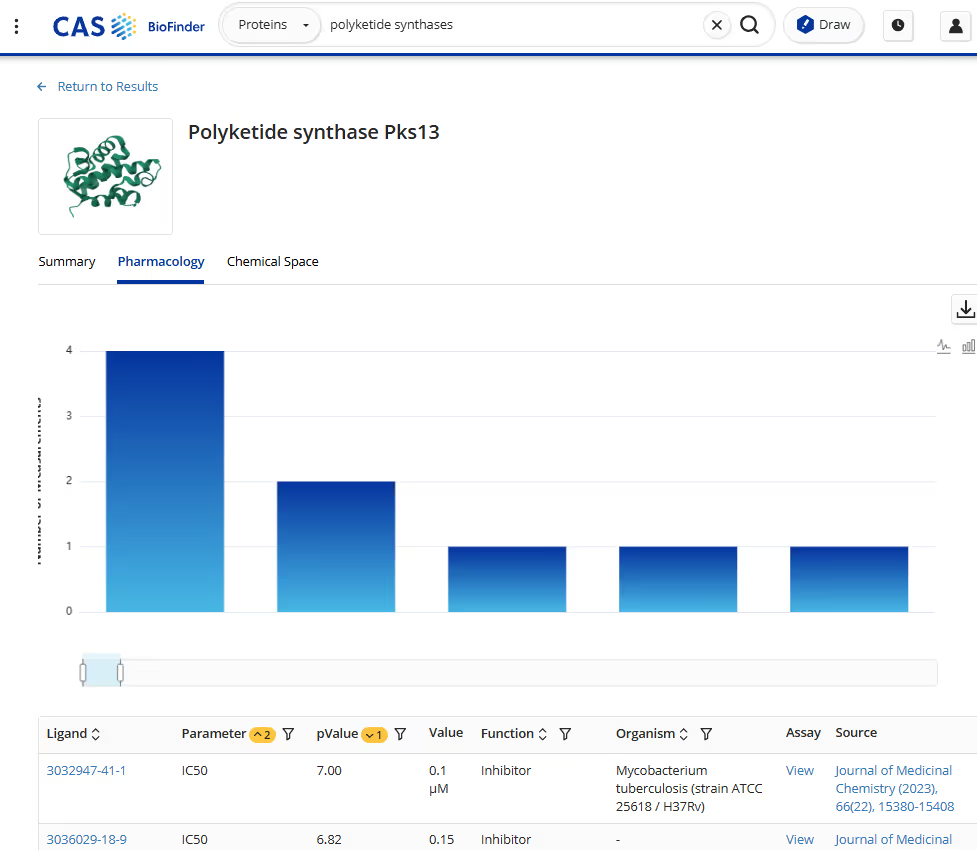
Outra área em que a IA está transformando nossa abordagem é a promiscuidade e especificidade das enzimas. As enzimas são naturalmente seletivas para seus substratos, mas muitas vezes precisamos que elas trabalhem com substratos não naturais para produzir novos derivados. Os modelos de IA podem nos ajudar a prever quais mutações ampliarão a atividade das enzimas sem sacrificar a eficiência.
CAS: Quais são alguns dos maiores gargalos relacionados a dados que você enfrentou em seu trabalho?
O maior desafio é a qualidade e a padronização dos dados. Os modelos de IA são tão bons quanto os dados que fornecemos a eles e, historicamente, a pesquisa biológica não foi estruturada para aprendizado de máquina em grande escala. Dados experimentais geralmente são armazenados em formatos diferentes, não possuem metadados ou não são bem anotados, o que dificulta sua integração e análise em escala.
Atualmente, muito do meu tempo é gasto organizando e limpando dados. Uma estrutura de dados unificada para dados biológicos, de quimionformática e gerados por IA aceleraria significativamente as descobertas.
Outro problema é a instrumentação e a extração de dados. Muitas de nossas técnicas analíticas (seja LC-MS, HPLC ou GC-MS) ainda exigem processamento manual de dados. Os fabricantes de instrumentos geralmente usam formatos proprietários que dificultam a extração e a análise automática dos resultados. Se tivéssemos formatos de dados padronizados e legíveis por máquina, poderíamos automatizar uma parte considerável do fluxo de trabalho, permitindo que os modelos de IA aprendessem diretamente com os resultados experimentais.
CAS: A colaboração entre pesquisadores de diferentes formações está se tornando essencial?
Em Berkeley, vemos em primeira mão como a colaboração interdisciplinar pode ser confusa, mas valiosa. Nosso departamento de bioengenharia inclui pessoas de biologia sintética, ciência da computação, engenharia de hardware e microfluídica. Essa diversidade nos permite lidar com problemas complexos que nenhuma disciplina única poderia resolver sozinha.
Uma área em que essa colaboração é fundamental é a ampliação da engenharia orientada por IA. Por exemplo, no JBEI, nosso laboratório de microfluídica está desenvolvendo novos dispositivos para otimizar a engenharia de cepas de alto rendimento. Seu trabalho nos permite testar milhares de projetos de proteínas diferentes em ciclos de engenharia que podem ser tão curtos quanto algumas semanas.
O desafio é que a pesquisa tem sido historicamente compartimentada. Químicos, biólogos e cientistas da computação frequentemente operam em esferas separadas, usando terminologia e estruturas diferentes. Precisamos de melhores plataformas de comunicação e conhecimento compartilhado para realmente integrar esses campos. Isso, por si só, aceleraria significativamente o progresso.
CAS: Se você tivesse uma varinha mágica para mudar algo no processo de descoberta de medicamentos, o que mudaria?
Minha resposta é um pouco filosófica, mas eu mudaria a forma como a pesquisa científica é valorizada e financiada — especialmente em biotecnologia. Atualmente, as startups e os laboratórios acadêmicos operam em um modelo de alto risco e alta recompensa, no qual muitas ideias promissoras nunca chegam a ser totalmente desenvolvidas porque não se encaixam nas estruturas tradicionais de financiamento. Se quisermos liberar o potencial da ciência de ponta, precisamos de investimentos de longo prazo em pesquisas fundamentais, não apenas em empreendimentos de curto prazo com fins lucrativos.
Eu criaria um sistema em que todas as pesquisas anteriores fossem instantaneamente acessíveis em um banco de dados estruturado e legível por máquina. O maior obstáculo que enfrentamos é que muitos dados valiosos estão trancados em PDFs e espalhados por diferentes repositórios. Se pudéssemos extrair e analisar instantaneamente tudo o que foi publicado sobre uma determinada via, isso aceleraria exponencialmente as descobertas.
Graham Hudson, Doutor, é bacharel em bioquímica pela Saint Louis University, onde pesquisou a termodinâmica de pares de bases de RNA para dar apoio ao projeto terapêutico. Concluiu seu doutorado na University of Illinois, em Urbana-Champaign, com Douglas A. Mitchell, concentrando-se em produtos naturais RiPP — reconstituindo a biossíntese de tiopeptídeos, descobrindo duas novas classes (rantipeptídeos e piritideos) e revelando a base enzimática da tioamidação. Atualmente, é pesquisador de pós-doutorado na UC Berkeley em Berkeley, no laboratório de Jay D. Keasling, e estuda a enzimologia e a biossíntese de produtos naturais de saponina. Ele também prestou consultoria para o setor em pesquisa de produtos naturais.
Nathan Lanclos é bacharel em biologia molecular e em economia pela University of South Florida, onde pesquisou a função da proteína no câncer com Vladimir Uversky e projetou o metabolismo de C1 em bactérias com Ramon Gonzalez. Atualmente, é estudante de doutorado no Programa Conjunto de Bioengenharia da UCSF/UC Berkeley. Em 2022, fundou uma empresa de consultoria que dá suporte a startups de biotecnologia com estratégia de marketing e modelagem de dados. Sua pesquisa se concentra em aprendizado de máquina para projeto de proteínas e engenharia de proteínas de alto rendimento, com ênfase em complexos de vários domínios, como sintases de policetídeos (PKSs) para produção química e terapêutica.



