
Los límites de la química sintética se están expandiendo al máximo, llevando a los investigadores a buscar soluciones en la biología. Con el auge de la biología sintética y la inteligencia artificial (IA), los científicos están descubriendo nuevas posibilidades para el descubrimiento de fármacos, como el diseño y la producción de moléculas raras y completamente nuevas en la naturaleza. Aprovechando estas herramientas, los investigadores están desarrollando enfoques innovadores para sintetizar moléculas complejas, optimizar rutas de producción y diseñar sistemas biológicos para la innovación farmacéutica. Para conocer mejor este campo en rápida evolución, hemos conversado con Graham Hudson, doctor en Biología Sintética y Microbiología, y con Nathan Lanclos, experto en diseño de proteínas basado en IA. Obtenga más información sobre cómo aborda CAS estos desafíos.
CAS: ¿Es la biología sintética el siguiente paso necesario para la innovación en el desarrollo de fármacos más allá de la química sintética?
En teoría, siempre que una estructura sea químicamente viable, la química sintética puede acceder a ella. Pero la cuestión no es únicamente la viabilidad, sino también la eficiencia, el costo y la escalabilidad. Existen compuestos que, teóricamente, podrían sintetizarse, pero el esfuerzo requerido para desarrollar una ruta y la enorme dificultad para controlar la selectividad los vuelve impracticables. Ahí es donde la biología sintética puede realmente ofrecer una nueva vía para la síntesis de fármacos.
La naturaleza ha dedicado millones de años a perfeccionar la maquinaria enzimática capaz de construir estas moléculas complejas con precisión extraordinaria, logrando a menudo una regio y estereoselectividad que puede ser extremadamente difícil de alcanzar con la química sintética. Si tomamos como ejemplo el paclitaxel, una molécula densa en centros estereogénicos y grupos funcionalmente similares, resulta extremadamente desafiante manipularla químicamente con la precisión necesaria para garantizar la formación del producto correcto, pero al aprovechar las herramientas biosintéticas de la naturaleza, podemos beneficiarnos de las enzimas que ensamblan estas moléculas de manera eficiente.
Además, podemos ir más allá con el auge de la biología computacional y el aprendizaje automático. Las herramientas de inteligencia artificial nos permiten diseñar enzimas que van más allá de lo que ha producido la evolución, modificando sus estructuras para catalizar reacciones novedosas o expandiendo su rango de sustratos. Esto significa que no solo estamos recreando productos naturales, sino que estamos abriendo la puerta a moléculas completamente nuevas para la naturaleza que podrían tener propiedades biológicas totalmente novedosas.
CAS: ¿Qué avances recientes están ayudando a los científicos a diseñar microbios para que sean «fábricas de medicamentos vivientes»?
Microbios como Saccharomyces (levadura) y Streptomyces (bacteria) ya se utilizan ampliamente para producir compuestos valiosos, desde antibióticos hasta agentes anticancerígenos.El principal desafío ha sido optimizar estos sistemas para producir altos rendimientos de manera eficiente. Los avances en IA, cribado de alto rendimiento y modelado computacional nos permiten diseñar y optimizar rutas biosintéticas a una escala sin precedentes.
Ahora podemos utilizar el aprendizaje automático para predecir cómo se comportarán las rutas metabólicas, lo que nos permite ajustar los niveles de expresión de las enzimas, optimizar la disponibilidad de precursores e incluso modificar el metabolismo del huésped para maximizar la producción. Por ejemplo, durante mucho tiempo ha sido difícil diseñar experimentos con grandes complejos proteicos heteroméricos para comprender en profundidad las interacciones proteína-proteína de largo alcance, pero ahora estamos encontrando formas de hacerlo con una resolución cada vez mayor.
Un ejemplo perfecto de cómo la biología sintética está transformando la producción de medicamentos es el QS-21 (Stimulon, abajo), un adyuvante inmunoestimulador utilizado en vacunas. Históricamente, el QS-21 se ha extraído del árbol de corteza de jabón (Quillaja saponaria), pero el proceso presenta varios desafíos importantes. El rendimiento de estos árboles es extremadamente bajo, lo que hace que la producción a gran escala sea insostenible ambientalmente e impracticable económicamente. Además, depender de la extracción vegetal significa que las cadenas de suministro son vulnerables a la variabilidad climática, la deforestación y factores geopolíticos.
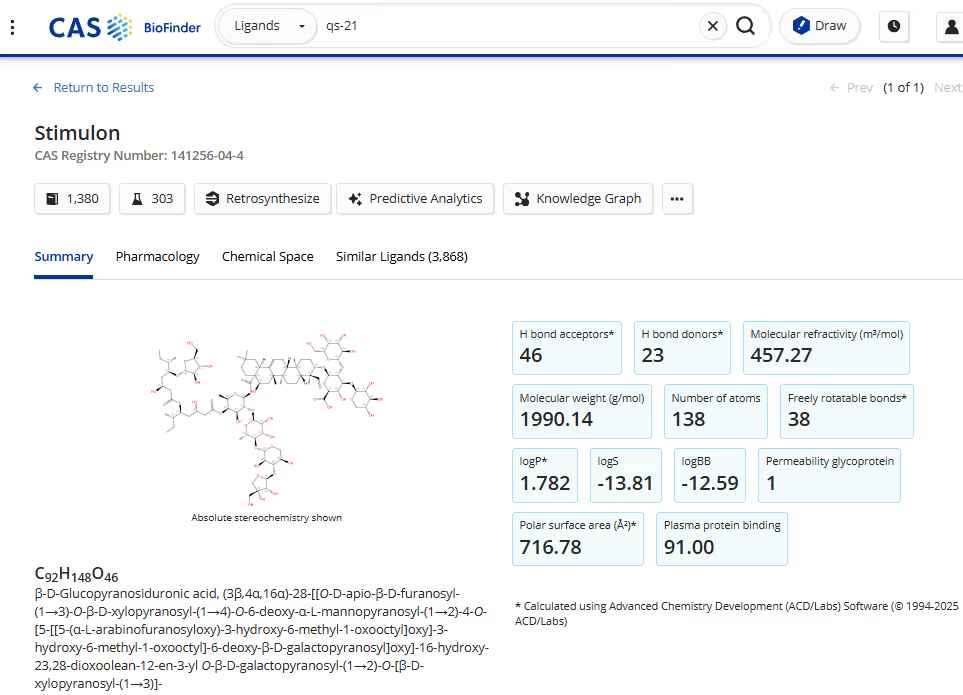
Antes de siquiera intentar diseñar una levadura para producir QS-21, primero tuvimos que mapear toda su ruta biosintética e identificar los genes responsables de su síntesis. Este proceso fue un esfuerzo hercúleo liderado por el laboratorio de Anne Osbourn en el John Innes Centre, que involucró un meticuloso análisis transcriptómico en diferentes etapas del ciclo de crecimiento de la planta.
A diferencia de los clústeres de genes biosintéticos microbianos, que tienden a estar localizados en el genoma, las rutas de las plantas están dispersas a través de múltiples cromosomas, a menudo con secuencias no codificantes entre los genes. Esta dispersión hizo que el descubrimiento de la ruta fuera mucho más complejo, requiriendo meticulosos estudios de correlación transcriptómica para determinar qué genes participaban funcionalmente en la biosíntesis del QS-21.
CAS: ¿Cuáles fueron los aspectos más complicados de adaptar las rutas biosintéticas derivadas de plantas para la producción microbiana?
Uno de los mayores desafíos fue la biosíntesis de azúcares. El QS-21 forma parte de la clase de productos naturales denominados saponinas, que son triterpenoides glicosilados complejos. Tiene siete azúcares diferentes unidos a su estructura central, pero la levadura solo produce uno de ellos de forma natural. Esto significaba que, además de introducir la ruta biosintética del QS-21, también teníamos que modificar genéticamente la levadura para sintetizar azúcares no nativos.
Otra cuestión importante era la toxicidad. Las saponinas como el QS-21 son compuestos activos en membranas que pueden alterar las bicapas lipídicas y secuestrar esteroles nativos críticos, lo que, en parte, es lo que los convierte en adyuvantes eficaces. Sin embargo, esto creó un problema considerable en la levadura: las moléculas de QS-21 comprometían la integridad de la membrana celular de la levadura, causando muerte celular y falla en la producción. Para resolver esto, tuvimos que modificar la composición de esteroles de la levadura para prevenir la alteración de la membrana y optimizar las condiciones de producción.
Todo este esfuerzo demuestra el poder de la biología sintética. Sin estos avances, la producción de QS-21 seguiría estando limitada por la extracción vegetal. Con cepas de bioproducción genéticamente modificadas, ahora tenemos una forma escalable, controlada y sostenible de producir QS-21. Aunque se requiere optimización adicional antes de que la producción a gran escala sea viable, este trabajo allana el camino para una formulación de vacunas más eficiente.
CAS: ¿En qué otras áreas está aprovechando la IA para acelerar el descubrimiento?
La IA nos permite diseñar y optimizar rutas biosintéticas de una manera que sería imposible únicamente con métodos tradicionales. En lugar de ensayo y error, podemos utilizar modelos predictivos para determinar qué modificaciones enzimáticas mejorarán la actividad, qué cepas huésped serán más compatibles y cómo interactuarán los diferentes componentes de la ruta.
Por ejemplo, en el laboratorio Keasling utilizamos IA para diseñar policétido sintasas (PKS), que producen una amplia gama de compuestos bioactivos, desde antibióticos hasta fármacos anticancerígenos. Las enzimas PKS son enormes, con módulos individuales que contienen miles de aminoácidos. Su inmenso tamaño y complejidad hacen que los enfoques tradicionales de ingeniería de proteínas sean extremadamente difíciles. La IA nos permite modelar estas proteínas gigantes, predecir cómo las modificaciones afectan la función y diseñar nuevas variantes de enzimas con actividad mejorada.
Otra área donde la IA está transformando nuestro enfoque es la promiscuidad y especificidad enzimática. Las enzimas son naturalmente selectivas respecto a sus sustratos, pero a menudo necesitamos que actúen sobre sustratos no naturales para producir derivados novedosos. Los modelos de IA pueden ayudarnos a predecir qué mutaciones ampliarán la actividad enzimática sin sacrificar la eficiencia.
CAS: ¿Cuáles son algunos de los mayores obstáculos relacionados con datos que han experimentado en su trabajo?
El mayor desafío es la calidad y estandarización de los datos. Los modelos de IA son tan buenos como los datos que les proporcionamos y, históricamente, la investigación biológica no se ha estructurado para el aprendizaje automático a gran escala. Los datos experimentales a menudo se almacenan en diferentes formatos, carecen de metadatos o no están bien anotados, lo que dificulta su integración y análisis a gran escala.
Actualmente, dedico demasiado tiempo a organizar y limpiar datos. Una estructura de datos unificada para datos biológicos, quimioinformáticos y generados por IA aceleraría significativamente los descubrimientos.
Otra cuestión es la instrumentación y extracción de datos. Muchas de nuestras técnicas analíticas, ya sean LC-MS, HPLC o GC-MS, aún requieren procesamiento manual de datos. Los fabricantes de instrumentos a menudo utilizan formatos propietarios que dificultan la extracción y análisis automatizado de resultados. Si tuviéramos formatos de datos estandarizados y legibles por máquina, podríamos automatizar una parte considerable del flujo de trabajo, permitiendo que los modelos de IA aprendan directamente de los resultados experimentales.
CAS: ¿Se está volviendo esencial la colaboración entre investigadores de diferentes campos?
En Berkeley, vemos de primera mano lo compleja, pero valiosa, que puede ser la colaboración interdisciplinar. Nuestro departamento de bioingeniería incluye personas de biología sintética, informática, ingeniería de hardware y microfluídica. Esta diversidad nos permite abordar problemas complejos que ninguna disciplina podría resolver por sí sola.
Un área donde esta colaboración es fundamental es la escalabilidad de la ingeniería impulsada por IA. Por ejemplo, en el JBEI, nuestro laboratorio de microfluídica está desarrollando dispositivos innovadores para optimizar la ingeniería de cepas de alto rendimiento. Su trabajo nos permite probar miles de diseños de proteínas diferentes en ciclos de ingeniería que pueden durar tan solo unas semanas.
El desafío es que, históricamente, la investigación ha estado compartimentalizada. Los químicos, biólogos e informáticos a menudo trabajan en ámbitos separados, utilizando terminología y marcos diferentes. Necesitamos mejor comunicación y plataformas de conocimiento compartido para integrar verdaderamente estos campos. Solo eso aceleraría enormemente el progreso.
CAS: Si tuviera una varita mágica para cambiar algo en el proceso de descubrimiento de fármacos, ¿qué cambiaría?
Mi respuesta es un poco filosófica, pero cambiaría la forma en que se valora y financia la investigación científica, especialmente en biotecnología. Actualmente, las startups y los laboratorios académicos operan bajo un modelo de alto riesgo y alta recompensa, donde muchas ideas prometedoras nunca se desarrollan completamente porque no encajan en las estructuras de financiamiento tradicionales. Si queremos liberar el potencial de la ciencia de vanguardia, necesitamos inversión a largo plazo en investigación fundamental, no solo en empresas impulsadas por ganancias a corto plazo.
Crearía un sistema donde toda la investigación previa fuera instantáneamente accesible en una base de datos estructurada y legible por máquina. El mayor obstáculo que enfrentamos es que gran parte de los datos valiosos están bloqueados en archivos PDF y dispersos a través de diferentes repositorios. Si pudiéramos extraer y analizar instantáneamente todo lo que se ha publicado sobre una ruta determinada, se aceleraría exponencialmente el descubrimiento.
Graham Hudson, doctor, obtuvo su licenciatura en Bioquímica en la Universidad de Saint Louis, donde investigó la termodinámica de los pares de bases del ARN para apoyar el diseño terapéutico. Completó su doctorado en la Universidad de Illinois en Urbana-Champaign bajo la dirección de Douglas A. Mitchell, especializándose en productos naturales RiPP: reconstituyó la biosíntesis de tiopéptidos, descubrió dos nuevas clases (rantipéptidos y pirítidos) y reveló las bases enzimáticas de la tioamidación. Actualmente es investigador postdoctoral en la Universidad de California en Berkeley, en el laboratorio de Jay D. Keasling, donde estudia la enzimología y biosíntesis de productos naturales con saponinas. También ha sido consultor para la industria en materia de investigación de productos naturales.
Nathan Lanclos es licenciado en Biología Molecular y Economía por la Universidad del Sur de Florida, donde investigó la función de proteínas en el cáncer con Vladimir Uversky y diseñó metabolismo C1 en bacterias con Ramón González. Actualmente es estudiante de doctorado en el Programa Conjunto de Bioingeniería UCSF/UC Berkeley. En 2022, fundó una empresa consultora que apoya a startups de biotecnología con estrategias de marketing y modelado de datos. Su investigación se centra en el aprendizaje automático para el diseño de proteínas y la ingeniería de proteínas de alto rendimiento, con énfasis especial en complejos multidominio como las policétido sintasas (PKS) para producción química y terapéutica.



