Desde que a Organização Mundial da Saúde declarou a Covid-19 uma pandemia, os pesquisadores aprenderam muito sobre o SARS-CoV-2, o novo coronavírus que causa essa doença. No entanto, apesar do extenso esforço e do alto investimento, tratamentos terapêuticos eficazes para pacientes com Covid-19 ainda são incertos. Embora várias vacinas candidatas já tenham entrado em ensaios clínicos em todo o mundo, mesmo que se mostrem seguras e eficazes, serão necessários muitos meses ou até anos para fabricar e distribuir a vacina e inocular a população global. Assim, permanece uma necessidade urgente de identificar tratamentos antivirais eficazes que possam mitigar o impacto do vírus em muitos outros que ficarão doentes antes que a pandemia seja controlada.
Os cientistas vêm explorando várias maneiras de acelerar o processo de desenvolvimento de medicamentos para atender a essa necessidade urgente, incluindo o uso de abordagens computacionais para identificar medicamentos já aprovados para outras indicações que possam ser eficazes no tratamento da Covid-19. Para ajudar nesse esforço, um grupo de cientistas e tecnólogos do CAS procurou identificar possíveis candidatos a medicamentos para o tratamento da Covid-19 com modelos de aprendizado de máquina para alvos proteicos prioritários do SARS-CoV-2 usando uma metodologia QSAR (Quantitative Structure-Activity Relationship). Este trabalho identificou com sucesso uma série de medicamentos que agora começam a mostrar eficácia clínica, incluindo Lopinavir e Telmisartan, foi publicado recentemente na ACS Omega.
O velho novo
Dado o tempo e o custo substanciais necessários para disponibilizar um novo medicamento ao mercado, o reaproveitamento de medicamentos de pequenas moléculas existentes é uma alternativa atraente, especialmente quando a necessidade é tão urgente. Além de disponibilizar os tratamentos ao mercado mais rapidamente, essa estratégia oferece várias vantagens em relação ao processo tradicional de desenvolvimento de medicamentos, incluindo a redução do risco de falha em estágio avançado devido a efeitos colaterais negativos.
O reaproveitamento de medicamentos não é um conceito novo. No entanto, sua aplicação até o momento tem sido mais oportunista do que sistemática. Em alguns dos exemplos mais bem-sucedidos de reaproveitamento de medicamentos até agora, como Viagra e Minoxidil, novas indicações surgiram quando os pacientes relataram efeitos colaterais inesperados. Recentemente, foram introduzidas abordagens mais sistemáticas para o reaproveitamento de medicamentos, incluindo métodos computacionais, como correspondência de assinatura, docking molecular, associação genética, mapeamento de vias e análise clínica retrospectiva. Espera-se que uma abordagem computacional permita que os pesquisadores conectem de forma confiável a terapêutica existente de pequenas moléculas a alvos de drogas recém-identificados, maximizando o valor terapêutico dos portfólios existentes.
Aproximando-se de um alvo
Os coronavírus são uma grande família de vírus há muito conhecida por causar doenças respiratórias leves a moderadas em humanos e em muitas outras espécies diferentes de animais. Embora seja raro que os coronavírus específicos de animais infectem e se espalhem em humanos, até o momento três coronavírus provaram ser capazes de dar esse salto: SARS-CoV-1, MERS-CoV e o novo SARS-CoV-2. Todos os três são beta-coronavírus que, se acredita, foram originados em morcegos. Dadas as semelhanças entre esses vírus e seu avanço para o contágio humano, pesquisas anteriores sobre SARS e MERS fornecem um bom ponto de partida ao buscar alvos farmacológicos para o SARS-CoV-2. Entre todas as proteínas do SARS-CoV-2, a protease do tipo 3-quimotripsina (3CLpro) e a RNA polimerase dependente de RNA (RdRp) são dois alvos proteicos ideais para modelagem de QSAR, em parte devido às semelhanças significativas que compartilham com proteínas identificadas no SARS-CoV e MERS-CoV, bem como outros coronavírus conhecidos.
A 3CLpro é uma protease necessária para o coronavírus clivar os peptídeos poliproteicos em proteínas não estruturais (PNEs) funcionais individuais. Ao comparar sequências de aminoácidos e estruturas de proteínas, verificou-se que o 3CLpro é altamente conservado entre o SARS-CoV-2 e outros coronavírus humanos. Ele mostra uma sobreposição de identidade de sequência de 96% com o SARS-CoV-1, 87% com o MERS-CoV e 90% com Coronavírus humano. Portanto, os inibidores 3CLpro identificados em pesquisas anteriores relacionadas ao coronavírus são inibidores promissores para o SARS-CoV-2 O 3CLpro, e os dados de relação estrutura-atividade (SAR) associados são valiosos para treinar modelos de aprendizado de máquina em busca de novos inibidores de SARS-CoV- 2 3CLpro.
RdRp é a principal enzima utilizada por vírus de RNA para replicar genomas virais em células hospedeiras. O estudo estrutural e a análise de sequência do SARS-CoV-2 RdRp revelaram que essa enzima é muito semelhante à estrutura do SARS-CoV-1 RdRp e contém vários resíduos de aminoácidos importantes que são conservados na maioria dos RdRps virais, incluindo o HCV. Felizmente, vários RdRps virais têm sido amplamente estudados como inibidores de vírus RNA, especialmente em pesquisas relacionadas ao HCV. Portanto, os inibidores de RdRp existentes para os vírus de RNA, como o HCV, podem fornecer informações valiosas para o desenvolvimento de medicamentos para inibição de RdRp do SARS-CoV-2.
Priorizando terapias existentes com aprendizado de máquina
Modelos de aprendizado de máquina têm sido cada vez mais usados para facilitar a descoberta de medicamentos nos últimos anos. Especificamente, o QSAR é, com frequência, um dos primeiros passos no processo moderno de descoberta de medicamentos. Simplificando, os QSARs são modelos matemáticos que aproximam propriedades biológicas ou físico-químicas bastante complicadas de produtos químicos com base em medidas quantitativas de suas estruturas moleculares. Esses modelos matemáticos preditivos são usados para fazer a triagem de dados de estruturas químicas em grandes bancos para priorizar potenciais candidatos a medicamentos com maior probabilidade de serem ativos contra alvos identificados. Essa abordagem pressupõe que a atividade de uma substância química está diretamente relacionada à sua estrutura e, portanto, moléculas com características estruturais semelhantes exibirão propriedades físicas e/ou efeitos biológicos semelhantes.
Neste estudo, meus colegas e eu colaboramos de perto para construir modelos QSAR altamente preditivos para alvos de proteína 3CLpro e RdRp. A equipe, que contou cientistas computacionais e químicos, selecionou mais de 1.000 inibidores com dados de estrutura-bioatividade como moléculas de treinamento para os modelos. Coletamos dados dos estudos mais atuais de bioensaio do SARS-CoV-2, bem como estudos existentes com SARS-CoV-1, MERS-CoV e outros vírus relacionados no CAS Content Collection. Usando esses dados, aplicamos uma variedade de algoritmos de aprendizado de máquina para construir várias dezenas de modelos QSAR – selecionando dentre eles os modelos de desempenho mais forte – um direcionado ao 3CLpro e outro direcionado ao RdRp.
Leia o artigo completo do periódico QSAR machine learning models and their applications for identifying viral 3CLpro- and RdRp-targeting compounds as potential therapeutics for COVID-19 and related viral infections para ver todos os modelos testados e que possíveis candidatos chegaram ao topo.
Usamos os dois modelos QSAR resultantes para fazer a triagem de um grande conjunto de potenciais candidatos a medicamentos, incluindo 1.087 medicamentos já aprovados pela FDA, quase 50.000 substâncias do conjunto de dados de compostos de candidatos antivirais, o CAS covid-19, e ~ 113.000 substâncias com atividade farmacológica identificada ou um papel terapêutico indexado pelo CAS em documentos relacionados a SARS, MERS e Covid-19 publicados desde 2003. Ao modelar a atividade do inibidor de protease em função da estrutura da substância, identificamos alguns dos candidatos mais promissores entre as substâncias previstas para serem inibidores ativos 3CLpro e RdRp do coronavírus. Além disso, várias substâncias que nossos modelos preveem que inibirão 3CLpro ou RdRp no SARS-CoV-2 também identificaram anteriormente atividade terapêutica contra outras doenças que surgiram como fatores de risco para infecções mais graves pela Covid-19. Por exemplo, um candidato a antiviral contra a Covid-19 que também possui atividade conhecida contra doenças cardíacas, como o cloridrato de diltiazem (Cardizem), poderia fornecer um benefício duplo, em certos casos.
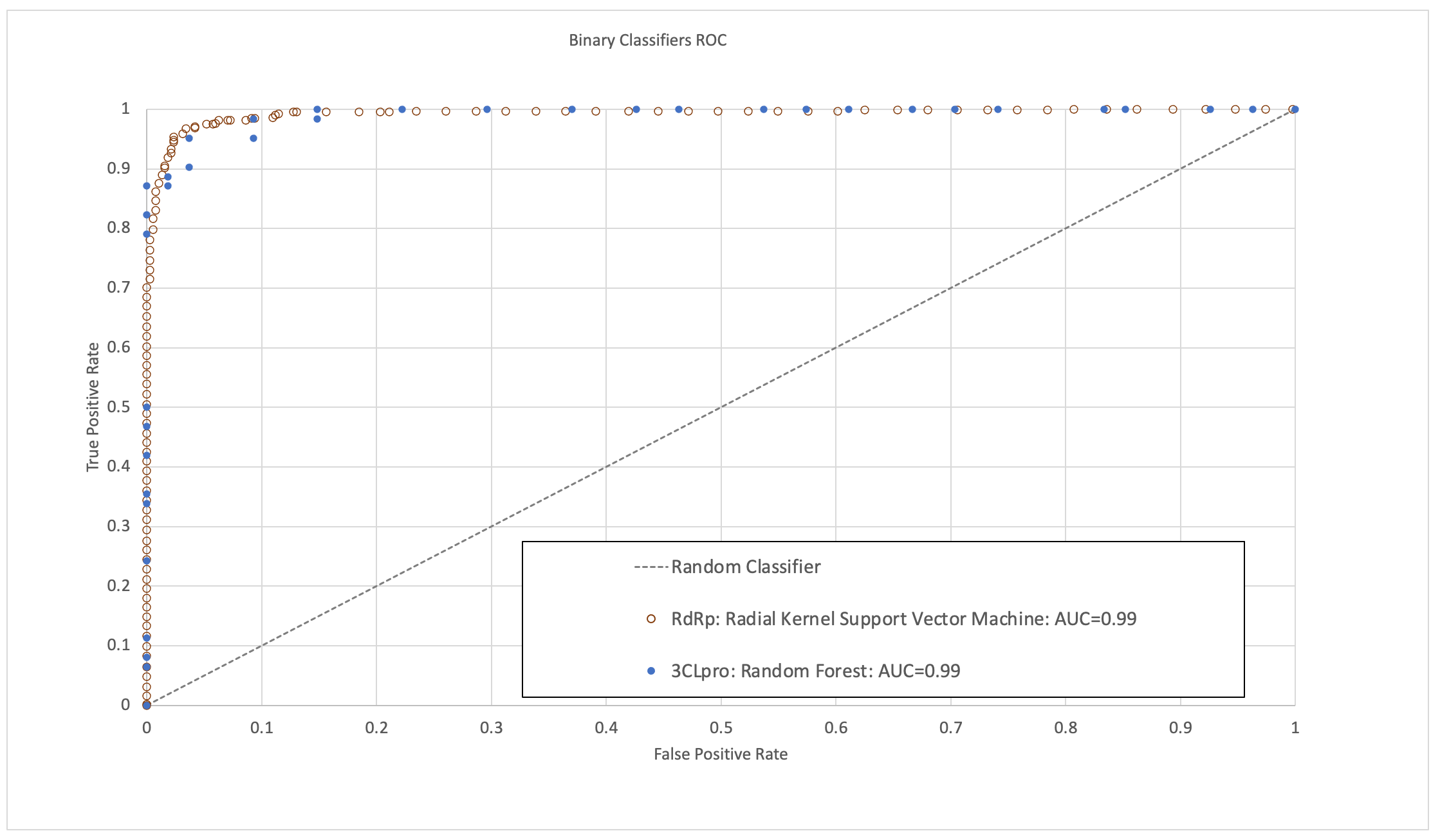
Os modelos foram validados para apresentar uma alta área sob a curva característica de operação do receptor (ROC-AUC), sensibilidade, especificidade e acurácia (Figura 1). No tempo desde que esta pesquisa foi concluída, algumas moléculas previstas para ter alta atividade por esses modelos já foram validadas por estudos de bioensaios experimentais publicados e estudos clínicos, fornecendo mais indicações positivas de sua capacidade preditiva.
Antecipando-nos à próxima pandemia
Embora este estudo tenha se concentrado na identificação de potenciais compostos terapêuticos para uso na atual crise da Covid-19, é provável que haja outras pandemias de origem viral nos próximos anos. Assim, é urgente começar agora a preparação para futuros surtos, investimento contínuo e foco na pesquisa de agentes antivirais. Como as epidemias podem ser causadas por diferentes tipos de vírus (por exemplo, coronavírus, vírus influenza, vírus Ebola, retrovírus) e os testes de segurança e eficácia em humanos para cada novo medicamento ou indicação ainda levam um tempo significativo, agentes antivirais de amplo espectro e vacinas seriam mais valiosos.
O docking molecular e triagem virtual serão de importância central no desenvolvimento contínuo de métodos de descoberta de medicamentos baseados em computador, como os procedimentos de aprendizado de máquina descritos aqui. O aumento contínuo do poder de processamento dos computadores e o desenvolvimento permanente de algoritmos de docking e previsão de estrutura e técnicas de determinação de estrutura de cristal de proteína facilitarão o avanço. Além disso, o uso de triagem de alto rendimento, tecnologias ômicas e o reaproveitamento de medicamentos já desenvolvidos continuarão e serão cada vez mais importantes. No entanto, esses novos métodos baseados em tecnologia não substituirão a pesquisa em laboratório feita por pessoas, mas serão uma complementarão por meio de maior eficiência. Esperamos que esse esforço, que combinou curadoria de dados feita por cientistas e modelos de aprendizado de máquina para identificar com sucesso potenciais candidatos a medicamentos de pequenas moléculas para a covid-19, destaque o valor da sinergia entre pessoas e máquinas na descoberta de medicamentos, ao mesmo tempo em que contribui com os esforços para a pesquisa antiviral em andamento para a covid-19 e muito mais.
Como parte da comunidade científica global, nós do CAS estamos comprometidos em alavancar todos os nossos ativos e recursos para apoiar a luta contra a Covid-19. Explore nosso CAS COVID-19 de acesso aberto, incluindo insights científicos, conjuntos de dados de acesso aberto e relatórios especiais.