
Desde que la Organización Mundial de la Salud declaró la pandemia de COVID-19, los investigadores han aprendido muchísimo sobre el SARS-CoV-2, el nuevo coronavirus causante de esta enfermedad. Sin embargo, a pesar de todos los esfuerzos y las inversiones, los tratamientos eficaces para los pacientes de COVID-19 han sido esquivos. Aunque ya se han iniciado ensayos clínicos para varias posibles vacunas en todo el mundo, incluso si se demostrase que son seguras y eficaces harían falta años para fabricarlas y distribuirlas e inocular a toda la población mundial. Por tanto, sigue siendo urgente identificar tratamientos antivirales eficaces que puedan mitigar el impacto del virus en los millones de personas que enfermarán antes de que se logre controlar la pandemia.
Los científicos están explorando varias formas de acelerar el proceso de descubrimiento de fármacos para responder a esta necesidad apremiante, incluido el uso de métodos computacionales para identificar medicamentos ya probados para otras patologías que puedan ser eficaces en el tratamiento de la COVID-19. Para contribuir a estos esfuerzos, un grupo de científicos y tecnólogos de CAS ha intentado identificar posibles fármacos para el tratamiento de la COVID-19 con la ayuda de modelos de aprendizaje automático para dianas proteínicas prioritarias del SARS-CoV-2 utilizando la metodología de relación cuantitativa estructura-actividad (QSAR, Quantitative Structure-Activity Relationship). Este trabajo, que ha logrado identificar varios fármacos que ahora están empezando a demostrar su eficacia clínica, como Lopinavir y Telmisartan, se ha publicado recientemente en ACS Omega.
Algo viejo, algo nuevo
Dada la gran cantidad de tiempo y dinero que se necesitan para llevar un nuevo medicamento al mercado, readaptar fármacos de moléculas pequeñas ya existentes es una alternativa atractiva, especialmente cuando la necesidad es tan urgente. Además de acortar los plazos de comercialización de los tratamientos, esta estrategia tiene algunas ventajas con respecto al proceso de desarrollo de fármacos tradicional, incluido un menor riesgo de fracaso en las últimas etapas del proceso a causa de la aparición de efectos secundarios negativos.
El reposicionamiento de fármacos no es un concepto nuevo. Sin embargo, su aplicación hasta la fecha ha sido más oportunista que sistemática. En algunos de los ejemplos de reposicionamiento de fármacos con más éxito documentados hasta la fecha, como los de Viagra y Minoxidil, las nuevas indicaciones se identificaron cuando los pacientes notificaron efectos secundarios no deseados. Recientemente se han introducido estrategias de reposicionamiento de fármacos más sistemáticas, entre las que se incluyen métodos computacionales como la comparación de firmas, el acoplamiento molecular, las asociación genética, el trazado de rutas y los análisis clínicos retrospectivos. Se cree que una estrategia computacional ayudará a los investigadores a conectar de manera fiable los tratamientos de moléculas pequeñas existentes con nuevas dianas farmacológicas para maximizar el valor terapéutico de las carteras de productos existentes.
Estrechar el cerco en torno a una diana
Los coronavirus son una extensa familia de virus conocidos desde hace tiempo por causar enfermedades de las vías respiratorias superiores entre leves y moderadas que afectan a los humanos y a muchas especies animales diferentes. Aunque es raro que los coronavirus específicos de animales infecten a humanos y se propaguen entre ellos, hasta la fecha hay tres coronavirus que han logrado dar el salto: el SARS-CoV-1, el MERS-CoV y el nuevo SARS-CoV-2. Los tres son betacoronavirus y se cree que se originaron en los murciélagos. Dadas las semejanzas entre estos tres virus y su salto al contagio humano, las investigaciones realizadas sobre el SARS y el MERS son un buen punto de partida para buscar posibles dianas para el tratamiento farmacológico del SARS-CoV-2. Entre todas las proteínas del SARS-CoV-2, la proteasa parecida a la 3-quimotripsina (3CLpro) y la ARN polimerasa dependiente de ARN (RdRp) son dos dianas proteínicas idóneas para la creación de modelos con QSAR, en parte por las numerosas similitudes que comparten con proteínas identificadas en el SARS-CoV y el MERS-CoV, así como en otros coronavirus conocidos.
3CLpro es una proteasa que se necesita para que el coronavirus escinda los péptidos de poliproteínas en proteínas no estructurales (NSP) funcionales individuales. Al comparar las secuencias de aminoácidos y las estructuras de las proteínas, se descubrió que 3CLpro presentaba un alto grado de semejanza entre el SARS-CoV-2 y otros coronavirus humanos. Muestra un solapamiento de identidad de la secuencia del 96 % con el SARS-CoV-1, un 87 % con el MERS-CoV y un 90 % con el Human-CoV. Por tanto, los inhibidores de la 3CLpro identificados en las investigaciones relacionadas con los coronavirus anteriores son prometedores para la proteasa 3CLpro del SARS-CoV-2, y los datos de la relación estructura-actividad (SAR) asociada son valiosos para entrenar modelos de aprendizaje automático para los nuevos inhibidores de la 3CLpro del SARS-CoV-2.
La RdRp es la principal enzima utilizada por los virus de ARN para replicar los genomas virales en las células del anfitrión. El estudio estructural y el análisis de secuencias de la RdRp del SARS-CoV-2 revelaron que esta enzima es muy similar a la estructura de la RdRp del SARS-CoV-1 y contiene varios residuos de aminoácidos esenciales que se conservan en la mayoría de las RdRp virales, como la del virus de la hepatitis C. Por suerte, varias RdRp virales se han estudiado ampliamente como inhibidores de los virus de ARN, especialmente en las investigaciones relacionadas con el virus de la hepatitis C. Por tanto, los inhibidores de RdRp existentes para los virus de ARN, como el de la hepatitis C, pueden proporcionar información valiosa para el desarrollo de fármacos para la inhibición de la RdRp del SARS-CoV-2.
El aprendizaje automático ayuda a priorizar los tratamientos existentes
En los últimos años, ha crecido el uso de los modelos de aprendizaje automático para facilitar el descubrimiento de fármacos. En concreto, la metodología QSAR es, con frecuencia, uno de los primeros pasos en el proceso moderno de descubrimiento de fármacos. En pocas palabras, las QSAR son modelos matemáticos que reproducen de manera aproximada propiedades biológicas o fisioquímicas bastante complicadas de sustancias químicas a partir de medidas cuantitativas de sus estructuras moleculares. Estos modelos matemáticos predictivos se usan para evaluar grandes bases de datos de estructuras químicas y priorizar los fármacos con más probabilidades de presentar actividad contra las dianas identificadas. Este método presupone que la actividad de una sustancia química está directamente relacionada con su estructura, de modo que las moléculas con características estructurales similares tendrán propiedades físicas y efectos biológicos similares.
En este estudio, mis colegas y yo colaboramos para crear modelos QSAR con una gran capacidad predictiva para las dianas proteínicas 3CLpro y RdRp. El equipo, integrado por científicos computacionales y químicos, seleccionó más de 1000 inhibidores con datos de estructura-bioactividad como moléculas de entrenamiento para los modelos. Exploramos la CAS Content Collection para recopilar datos de los estudios de bioanálisis más recientes del SARS-CoV-2, así como de estudios sobre el SARS-CoV-1, el MERS-CoV y otros virus relacionados. Usando estos datos, aplicamos un conjunto de algoritmos de aprendizaje automático para crear varias docenas de modelos de QSAR y seleccionamos los que ofrecían un rendimiento más alto, uno para 3CLpro y otro para RdRp.
Lea el artículo completo QSAR machine learning models and their applications for identifying viral 3CLpro- and RdRp-targeting compounds as potential therapeutics for COVID-19 and related viral infections para ver todos los modelos estudiados y descubrir los candidatos que presentaron los mejores resultados.
Usamos los dos modelos QSAR resultantes para evaluar un amplio grupo de fármacos candidatos, entre los que se incluyeron 1087 medicamentos aprobados por la FDA, casi 50 000 sustancias del Conjunto de datos de compuestos candidatos antivirales para la COVID-19 de CAS y unas 113 000 sustancias con actividad farmacológica identificada o con una función terapéutica indexada por CAS en documentos relacionados con el SARS, el MERS y la COVID-19 publicados desde 2003. Al crear un modelo de la actividad inhibidora de la proteasa como función de la estructura de la sustancia, identificamos algunos de los candidatos más prometedores entre las sustancias seleccionadas por su potencial para convertirse en inhibidores activos de la 3CLpro y la RdRp del coronavirus. Además, varias de las sustancias que, según nuestros modelos, inhibirán la 3CLpro o la RdRp en el SARS-CoV-2 también han presentado previamente actividad terapéutica contra otras enfermedades que se han identificado como factores de riesgo para las infecciones más graves de COVID-19. Por ejemplo, uno de los posibles antivirales para la COVID-19 que también tiene actividad conocida contra las enfermedades cardiacas, como el hidrocloruro de diltiazem (Cardizem), podría ser doblemente beneficioso en algunos casos.
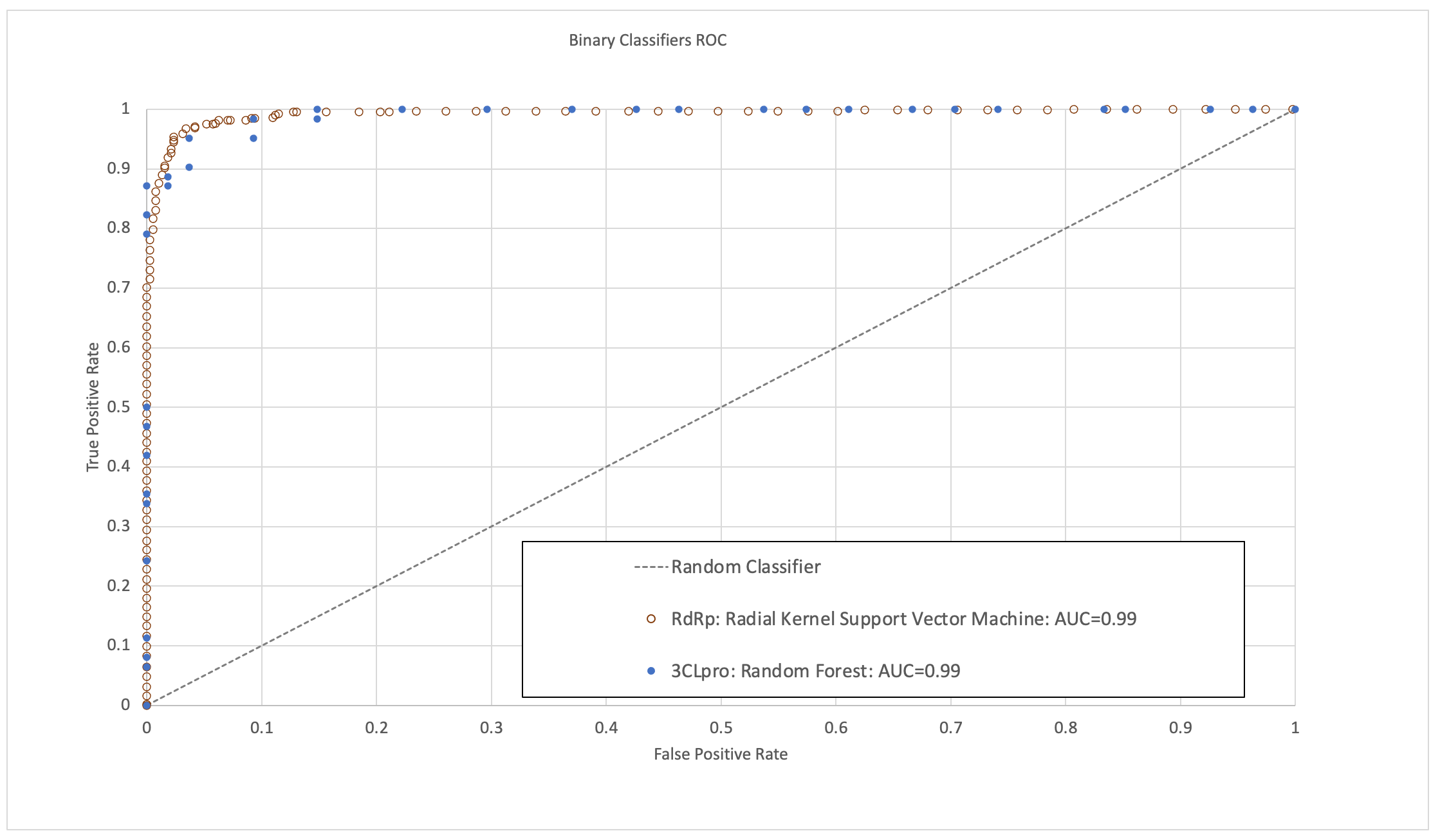
Los modelos se validaron de manera que presentaran un área grande debajo de la curva de característica operativa del receptor (ROC-AUC), sensibilidad, especificidad y precisión (figura 1). En el tiempo transcurrido desde que se completó esta investigación, algunas de las moléculas para las que estos modelos predijeron una actividad elevada se han validado mediante estudios de bioanálisis experimental y ensayos clínicos publicados, lo que confirma su capacidad predictiva.
Anticiparse a la próxima pandemia
Aunque este estudio se centró en la identificación de compuestos con potencial terapéutico para el uso en la crisis debida al COVID-19, es probable que en los próximos años surjan nuevas pandemias de origen vírico. Por este motivo es importante mantener la inversión y seguir investigando agentes antivirales para iniciar ya la preparación para los brotes futuros. Dado que hay distintos tipos de virus que pueden causar epidemias (p. ej., coronavirus, virus de la gripe, ébola, retrovirus) y puesto que las pruebas para verificar la eficacia y la seguridad en humanos de cada nuevo fármaco o cada nueva indicación requieren mucho tiempo, contar con vacunas y agentes antivirales de amplio espectro sería enormemente útil.
El desarrollo continuo de métodos de descubrimiento de fármacos computacionales, como los procedimientos de aprendizaje automático que se han descrito aquí, el acoplamiento molecular y el cribado virtual, será esencial. El aumento de la capacidad de procesamiento computacional y el desarrollo constante de algoritmos de predicción de estructuras y acoplamiento y de técnicas de determinación de la estructura cristalina de las proteínas facilitarán el progreso. Además, el uso del cribado de alto rendimiento, las tecnologías ómicas y el reposicionamiento de medicamentos seguirán avanzando y ganando protagonismo. Estos nuevos métodos tecnológicos no reemplazarán a la investigación en el laboratorio realizada por humanos, sino que la complementarán mejorando su eficiencia. Esperamos que esta iniciativa, en la que se combinó la selección de datos por expertos con modelos de aprendizaje automático para identificar fármacos de moléculas pequeñas que se puedan usar como tratamientos para la COVID-19, ponga de manifiesto el valor de la sinergia entre los humanos y las máquinas en el descubrimiento de fármacos y contribuya a los esfuerzos de investigación de antivirales para la COVID-19 y otras enfermedades que se están desarrollando actualmente.
Como miembro de la comunidad científica internacional, CAS ha puesto todos sus recursos y capacidades al servicio de la lucha contra la COVID-19. Explore los recursos adicionales de CAS para la COVID-19, que incluyen información científica, conjuntos de datos de acceso abierto e informes especiales.
